SARS-CoV-2 ist, nach SARS-CoV und MERS-CoV, ein weiterer zoonotischer Erreger aus der Gattung Betacoronaviren, der von Fledermäusen als Reservoirwirt erfolgreich Eingang in die menschliche Population gefunden hat. Die Rolle von potenziellen Zwischenwirten wie dem Schuppentier wird aktuell intensiv erforscht und ist noch nicht hinreichend geklärt.
Das von SARS-CoV-2 im Menschen induzierte Krankheitsgeschehen manifestiert sich als primär respiratorische Infektion und wurde von der WHO mit „COVID-19“ bezeichnet. Nach der Erstbeschreibung in China im Dezember 2019 verbreitete es sich innerhalb weniger Monate über die gesamte Welt mit über 100 Millionen labordiagnostisch bestätigten Infektionen und über 2,5 Millionen Toten (Statistik der WHO, Stand Februar 2021).
Das Infektionsgeschehen um SARS-CoV-2 dokumentiert eindrücklich, wie Forschung und Medizin in gemeinsamer globaler Anstrengung auf Pandemien antworten können. Sehr schnell wurden Gesamtgenomsequenzen zur Verbesserung der molekularen Diagnostik auf öffentlich zugänglichen Plattformen publiziert, Manifestationsformen des Krankheitsbildes bei verschiedenen Patientengruppen beschrieben und vielfältige Therapieregime auf ihre Wirksamkeit und ihren Nutzen hin untersucht.
Lebenszyklus von SARS-CoV-2
SARS-CoV-2 ist ein von einer Lipidmembran umhülltes Virus mit einem einzelsträngigen RNA-Genom von ca. 30 kB. Das auf der Partikeloberfläche befindliche virale Spike-Protein vermittelt die Bindung an empfängliche Wirtszellen hauptsächlich über das Protein ACE2 (angiotensin converting enzyme type 2) als Rezeptor. Über Clathrin-vermittelte Pinozytose werden SARS-CoV-2-Partikel mithilfe der Aktivität von Serinproteasen in die Zelle aufgenommen, der Replikationszyklus im Zytoplasma ausgelöst und reife Nachkommenviren durch Lyse der Zelle freigesetzt [1]. Das Ausmaß der Virusproduktion im Wirt ist abhängig von der Reaktion des Immunsystems und den Charakteristika verschiedener SARS-CoV-2-Stämme. Der Gewebetropismus erstreckt sich vor allem auf empfängliche Zelltypen des oberen und unteren Respirationstraktes [2]. Darüber hinaus wurde die Fähigkeit zur Replikation auch in einem großen Spektrum von Zelltypen anderer Gewebe nachgewiesen, unter anderem im Darm, im zentralen Nervensystem, in Leber, Niere, Herz und den Blutgefäßen (v. a. Endothelzellen).
Der Nachweis gelingt am besten aus Sputum und tiefen Nasopharynx-Abstrichen, wo SARS-CoV-2-Konzentrationen von bis zu 1012 Genomkopien/ml detektiert werden können [3, 4]. Virämien sind zwar dokumentiert (v. a. bei schweren Verläufen), jedoch nicht die Regel. Bei milden Verläufen ist nur sehr selten Virus im Blut nachweisbar [4, 5].
Immunreaktion gegen SARS-CoV-2
Die Ausprägung der Immunantwort des Wirtes mit Auswirkungen auf Krankheitsverlauf und -schwere ist abhängig von zahlreichen Faktoren des Wirts wie Alter, Geschlecht, Ethnie und T-Lymphozyten-Funktion sowie vom Virusstamm und dessen Charakteristika (Infektiosität, Pathogenität). Je jünger ein Infizierter, umso besser ist dessen Immunreaktion, die sich in ausreichend hohen, gut neutralisierenden IgG-Konzentrationen messen lässt und in einer effektiven T-Zellantwort mündet [6]. Bei älteren Infizierten ist die gebildete Anti-SARS-CoV-2-IgG-Konzentration höher als bei jüngeren, aber die neutralisierende Kapazität ist meist geringer und die T-Zellantwort weniger effektiv. Die messbaren Antikörper-Spiegel im Blut vermindern sich bei jungen Rekonvaleszenten nach ca. 3 Monaten, bei älteren erst nach ca. 6 Monaten. Männer neigen im Vergleich zu Frauen zur Bildung höherer Antikörper-Spiegel [7]. Wie dies im Zusammenhang mit der tendenziell höheren Sterblichkeit von Männern bei COVID-19 im Gegensatz zu Frauen steht [8], ist noch nicht geklärt. Da SARS-CoV-2 Makrophagen nicht effizient infiziert, sind sogenannte ADE-Effekte (antibody dependent enhancement) zu vernachlässigen [9].
Reinfektionen mit SARS-CoV-2 nach überstandener Erstinfektion sind mehrfach beschrieben worden. Typischerweise war in diesen Fällen die Erstinfektion mild und führte zu einer kaum messbaren IgG-Produktion. Die Zweitinfektion durch einen anderen SARS-CoV-2-Stamm kann von asymptomatisch bis schwer symptomatisch verlaufen und ist dann mit einer gut messbaren Immunantwort (IgG und IgM) verbunden [10]. Eine sehr starke Immunantwort kann die Pathogenität von SARS-CoV-2 erhöhen, weswegen am Anfang der Epidemie das verabreichte Hydroxychloroquin teils zu einer geringeren Mortalität führte [11].
Spezifische endemische Infektionsmuster sind bei vielen Coronaviren bekannt. Sie zeichnen sich durch eine Selektion hin zu niedrigpathogenen Virusstämmen mit einer effizienten Unterdrückung der Immunantwort aus. Infektionskreisläufe entstehen dann durch eine Reinfektion solcher Individuen mit schwindender Immunität, durch einen Eintrag antigenetisch anderer Virusstämme oder eine Infektion immunologisch naiver Individuen [12]. Dauerausscheider können grundsätzlich eine Rolle spielen, sind bei SARS-CoV-2 aber bisher nicht von Bedeutung.
SARS-CoV-2-Mutanten/-Varianten
SARS-CoV-2 ist als RNA-Virus einer ständigen Evolution unterworfen. Aufgrund der besonderen Biologie von Coronaviren mit einem außergewöhnlich großen Genom besitzen Vertreter dieser Familie in ihrem Replikationskomplex jedoch gewisse Korrekturmechanismen, die zum Erhalt des Genoms notwendig sind [13]. Coronaviren zeichnen sich deshalb grundsätzlich durch eine etwas niedrigere Mutationsneigung aus als andere RNA-Viren wie z. B. Influenzaviren. Dennoch ist das Auftreten neuer Varianten bei SARS-CoV-2 durch seinen Wirtswechsel und die weltweite Verbreitung in einer zuvor naiven Wirtspopulation vorhersagbar und auch dokumentiert.
Bereits einen Monat nach den ersten Meldungen des SARS-CoV-2-Ausbruchs in Wuhan tauchte in China und Taiwan ein Virus mit einer Deletion im ORF8 (Delta382) auf, was zum Funktionsverlust
es zugehörigen viralen Proteins führte [14]. Die Mutation Delta382 beeinträchtigt zwar nicht die Replikation des Virus, moduliert jedoch die Interaktion mit der Immunantwort des Wirtes. Das durch ORF8 kodierte virale Protein spielt eine Rolle in der Suppression der IFN-Antwort. Das Delta382-Virus führt damit zu einem milderen Verlauf der Infektion mit reduzierter Todesrate, aber dennoch zu einer messbaren Immunantwort [15].
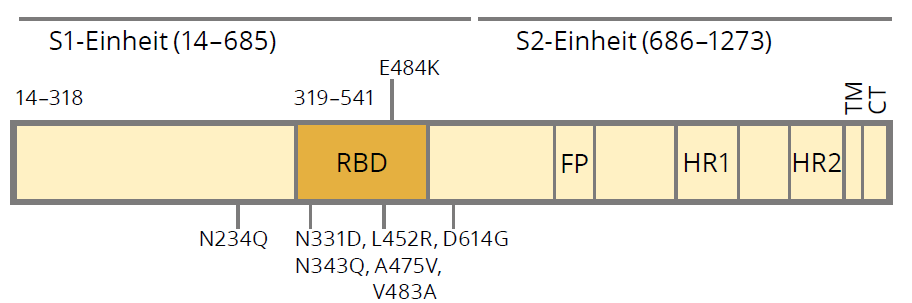
Eine weitere Variante mit bedeutender Verbreitung ist das D614G-Virus (Austausch von Asparaginsäure gegen Glycin im Spike-Protein). Durch diese Mutation in der RBD (Rezeptorbindedomäne) des Spike-Proteins weist es eine gesteigerte Infektiosität auf, was sich auch deutlich in einer hohen Kompetitivität und Verdrängung anderer Virusstämme wie dem Wildtyp auszeichnet [16]. Auswirkungen dieser Mutation auf die Pathogenität werden sehr kontrovers diskutiert und sind noch nicht abschließend geklärt.
Gegen Ende 2020 traten weitere Varianten und Mutationsmuster wie „B1.1.7“ oder das Nerz-assoziierte „Cluster 5“ auf, die ebenfalls eine hohe Kompetitivität und Neigung zum Verdrängen anderer Virusstämme besitzen. In Manaus, Brasilien, zirkuliert derzeit das sog P1-Virus, das wie die britische Variante D614G trägt. Weitere Mutationen wie etwa N501Y, E484K und D839Y scheinen eine leichtere Übertragbarkeit von Mensch zu Mensch und wohl auch schwerere Verläufe zu bedingen, denn P1 hat in Brasilien zu einer massiven zweiten Infektionswelle mit stark steigender Zahl an Infizierten und Toten geführt. Das Monitoring solcher ständig neu auftretenden Virusvarianten ist essenziell, um Veränderungen im Verhalten des Virus in Bezug auf die Epidemiologie, die Pathogenese und die Antigenität mit Auswirkung auf die Effizienz von Vakzinen beurteilen zu können. Nur mit diesem Wissen können im Bedarfsfall Interventionsmechanismen angepasst oder Impfstoffe moduliert werden.