Schlüsselwörter: Ringversuch, Faktor II, Faktor V, Protein C, Protein S, Antithrombin
Die Erforschung genetischer Ursachen der Gerinnungsstörungen bei Venenthrombosen und Lungenembolien hat gezeigt, dass Mutationen in fünf Proteinen zur Prädisposition der hereditären Thrombophilie beitragen: Bei einem Drittel der Thrombosepatienten unter 50 Jahren lässt sich eine Mutation im Faktor-V- bzw. im Prothrombin-Gen nachweisen. Mutationen der Gerinnungsinhibitoren Antithrombin, Protein C und Protein S treten seltener auf, sind aber mit einem höheren Erkrankungsrisiko verbunden.
Molekulargenetische Analysen, die den qualitativen Nachweis einer Mutation zum Ziel haben, werden häufig nur einmal im Leben eines Patienten durchgeführt. Daher ist es essenziell, dass die Ergebnisse dieser Untersuchungen verlässlich sind. Ringversuche bieten hier eine externe Qualitätskontrolle.
Faktor V
Die Aktivität des Prothrombinasekomplexes aus den aktivierten Faktoren V (FVa) und X (FXa) wird auch durch Protein C geregelt, das FVa proteolytisch spaltet. In die Jahre 1993/1994 fiel die Entdeckung einer Punkt-Mutation (single nucleotide polymorphism, SNP) des Faktors V, die dem aktivierten Faktor erhebliche Resistenz gegen die Inaktivierung durch aktiviertes Protein C (APC) verleiht [1, 2]. Bei der nach ihrem niederländischen Entdeckungsort Leiden benannten Mutation des Faktor-V-Gens (FVL) findet sich anstelle der Base Guanin jetzt Adenin (c.1691G>A). Dieser SNP hat zur Folge, dass in der Position 506 der Polypeptidkette die Aminosäure Arginin durch Glutamin ersetzt wird (p.Arg506Gln; gemäß der HGVS-Nomenklatur trägt die Mutation die Bezeichnung p.Arg534Gln). Die Spaltung durch APC hinter diesem Arginin ist Voraussetzung für die eigentliche Inaktivierung durch die Spaltung hinter den Argininresten 306 und 679 [3].
Die Träger der Faktor-V-Leiden-Mutation sind bevorzugt europäischer Abstammung – wenn auch mit erheblichen geografischen Unterschieden. In Deutschland beträgt die mittlere Prävalenz 5,9% – auch hier mit ausgeprägten regionalen Unterschieden [4].
Das Risiko für eine tiefe Venenthrombose korreliert mit dem Anteil der Faktor-V-Leiden-Proteine im Blut. So ist bei heterozygoten Erbmalsträgern die Wahrscheinlichkeit für ein Thromboseereignis drei- bis achtfach erhöht [5]. Dagegen besitzen homozygote Merkmalsträger des Faktor-V-Leiden eine wesentlich höhere Prävalenz. Einige Autoren finden ein bis zu achtzigfach erhöhtes Risiko [5, 6, 7]. In der westlichen Bevölkerung ist die APC-Resistenz in mehr als 90% der Fälle eine Folge der FV-Leiden-Mutation.
Bei hetero- und homozygoten Merkmalsträgern besteht ein erhöhtes Risiko für venöse Thrombembolie (VTE), mit einem Quotenverhältnis (Odd-Ratio) von 3,5 bzw. 18 (bezogen auf Familienmitglieder ohne bzw. mit FVL-Mutation).
Faktor II (Prothrombin)
Im Jahr 1996 wurde eine Mutation in der Position 20210 auf Chromosom 11 entdeckt, die zur 3‘ UT-Region des Prothrombingens gehört. Bei diesem SNP in der nicht-translatierten Region ist die Base Guanin gegen Adenin ausgetauscht (c:20210G>A). Bei Merkmalsträgern ist der Plasmaspiegel von Faktor II leicht erhöht [8]. Dieser Polymorphismus fand sich in 18% der Probanden aus Familien mit Thrombosen und nur in 2% von gesunden Kontrollpersonen. Die genaue molekulare Bedeutung dieser Mutation ist bislang unbekannt.
In Europa liegen die Häufigkeiten dieser Mutation zwischen 1% und 3% mit leicht höheren Anteilen in Südeuropa [9]. Für Personen mit dieser Mutation werden bei heterozygot vorliegendem Merkmal relative Thromboserisiken von 2 bis 8 gefunden [8, 10, 11]. Zwischen 3% und 8% der Patienten mit VTE besitzen dieses Merkmal, und für die Prävalenz eines Rezidivs werden 15% bis 20% angegeben [11].
INSTAND e. V. etablierte die Ringversuche zur Faktor-V-Leiden- und zur PT20210G>A-Mutation im Jahr 1999. Als Probenmaterial wurde zunächst ausschließlich isolierte DNA versendet. Seit 2006 werden für diese beiden Gerinnungsfaktoren auch Vollblutringversuche durchgeführt.
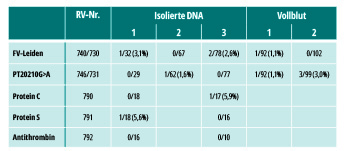
Für die Vollblutanalysen des ersten Ringversuchstermins 2017 ergab sich für FVL und PT20210G>A unter jeweils 92 Teilnehmern je ein Fehler (von verschiedenen Einsendern). Beim zweiten Ringversuchstermin mit ähnlicher Teilnehmerzahl waren für FVL alle Einsendungen richtig, aber drei Laboratorien fanden nicht die richtigen Genotypen für PT20210G>A.
An den Ringversuchen, bei denen isolierte DNA eingesetzt wurde, nahmen weniger Labore teil: In der ersten Serie war eine von insgesamt 32 Einsendungen fehlerhaft; in der dritten Serie zwei von 78. In beiden Ringversuchsserien waren alle Analysen für PT20210G>A richtig. In der zweiten Serie waren alle 67 Einsendungen für FVL richtig, aber eine unter 62 für PT20210G>A fehlerhaft.
Alle Fehler stammten aus verschiedenen Laboratorien. Keiner der Teilnehmer mit falschen Analyseergebnissen berichtete über eine mangelhafte Probenqualität. Die Erfolgsrate für 2017 ist repräsentativ für die Ergebnisse in anderen Jahren.
Die möglichen Fehlerquellen sind analytische Fehler, Verwechselungen bei der Protokollierung (intern oder bei der Übertragung in die Ringversuchsunterlagen) und Probenverwechselungen. Erfreulicherweise gibt es immer seltener Grund zu der Annahme, dass einer der beiden letzten Punkte eine Rolle spielt.