Leistungsfähiges Verfahren ohne Eingriffsrisiko
Nicht-invasive Pränataltestung (NIPT)
Die Einführung nicht-invasiver Verfahren in die pränatale Patientenversorgung stellt einen Paradigmenwechsel dar. Ohne eingriffsbedingtes Risiko erkennt man anhand der fetalen DNA im mütterlichen Blut die Trisomien 13, 18 und 21 sowie geschlechtschromosomale Aneuploidien. NGS-basierte Verfahren werden erläutert und diskutiert.
Schlüsselwörter: Nicht-invasive Pränataltestung, zellfreie, fetale DNA, fetale Fraktion, Trisomie
Das Risiko für chromosomale Fehlverteilungen des Fetus nimmt mit dem Alter der Schwangeren zu. Am häufigsten findet man die Trisomie 21 (Down-Syndrom), deren Inzidenz zwischen dem 20. und 50. Lebensjahr der Mutter von etwa 0,1% auf fast 10% ansteigt. Diese Feten weisen Fehlentwicklungen auf, sind aber über das dritte Trimester und die Geburt hinaus durchaus lebensfähig. Um Zehnerpotenzen seltener werden Kinder mit den Trisomien 13 (=Pätau-Syndrom) und 18 (Edwards-Syndrom) oder mit geschlechtschromosomalen Aneuploidien (X, XXY, XYY, XXX) geboren.
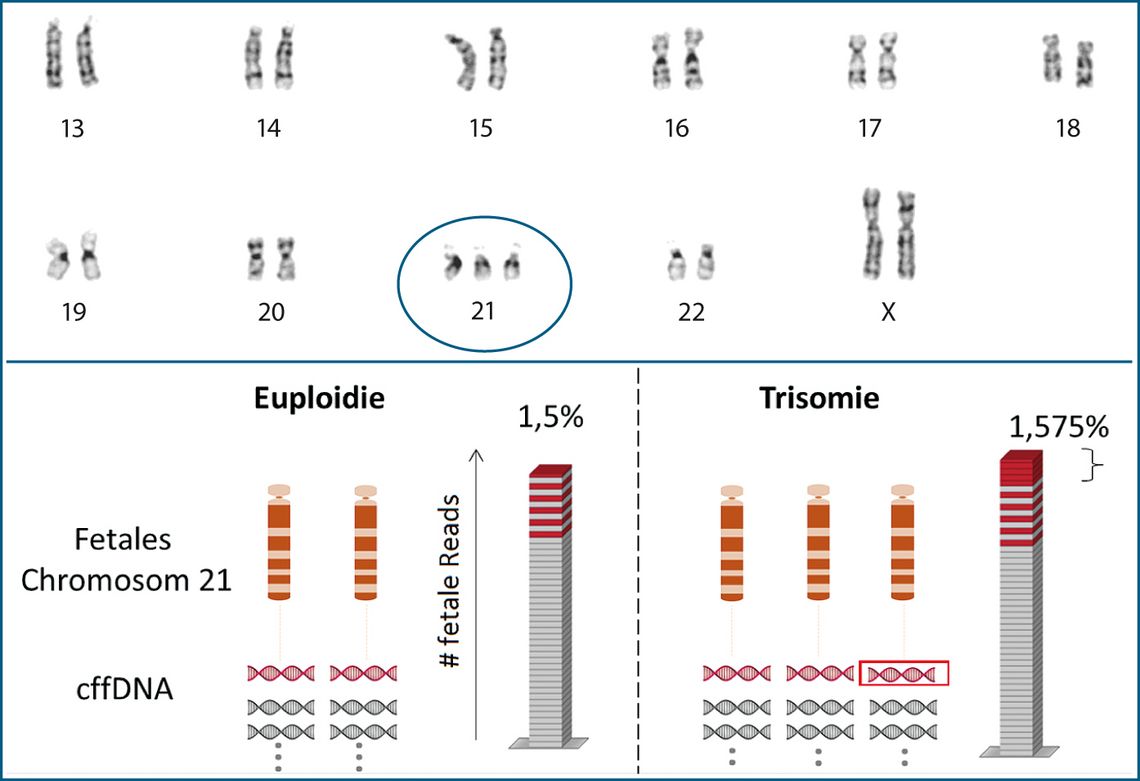
Mittels Ultraschalluntersuchung und Serumanalytik können im Rahmen des Erst- oder Zweittrimester-Screenings verschiedene Risikoindizes erfasst und bewertet werden. Deutet der Wert auf eine Trisomie hin, so können zwei invasive Verfahren Klarheit schaffen: die Fruchtwasseruntersuchung (Amniozentese, AC) und die Chorionzottenbiopsie (CVS). Aus dem Untersuchungsmaterial bestimmt man den Karyotyp mikroskopisch, und erhält so eine sehr sichere Aussage über Anzahl und Struktur der fetalen Chromosomen (Abb. 1 oben).
Nicht-invasive Verfahren
Das herkömmliche Screening liefert allerdings – nicht zuletzt aufgrund der geringen Inzidenz – mehr falsch- als richtig-positive Ergebnisse, und die invasiven Folgeuntersuchungen weisen ein Eingriffsrisiko von bis zu 0,2% auf. Mögliche Folgen wie Infektion, Fruchtwasserverlust oder im schlimmsten Falle Fehlgeburt müssen gegen das deutlich geringere Risiko einer chromosomalen Fehlverteilung abgewogen werden.
Die Suche nach risikoärmeren Screeningverfahren führte vor über 40 Jahren zur Entdeckung fetaler Zellen im mütterlichen Blut. Sie schienen zunächst für eine nicht-invasive Karyotypisierung bestens geeignet, erwiesen sich aber in der Praxis als problematisch, da sie in sehr geringer Zahl vorlagen und nur schwer spezifisch angereichert werden konnten[1]. Neue Hoffnung brachte die Entdeckung der zellfreien fetalen DNA (cffDNA) im Jahre 1997[2], die durchschnittlich 10% der gesamten zellfreien DNA (cfDNA) im mütterlichen Blut ausmacht. Ihre Fragmente repräsentieren das komplette kindliche Genom und sind äußerst stabil, da sie zum Schutz vor enzymatischem Abbau in Mikropartikel verpackt vorliegen.
Seit 2012 wird die nicht-invasive Pränataltestung auf Basis fetaler DNA-Fragmente in Deutschland als Screeningverfahren auch kommerziell angeboten. Das Konzept ist in Abb. 1 unten dargestellt. Ein Fetus bzw. seine Plazenta mit einem zusätzlichen Chromosom 21 (Trisomie 21) gibt im Vergleich zu einem nicht betroffenen Fetus eine zusätzliche Portion von Chromosom 21 spezifischer, fetaler cfDNA in das mütterliche Blut ab. Dieser kleine Mengenunterschied der cfDNA kann mithilfe der NGS-Technologie mit hoher Zuverlässigkeit detektiert werden. Dabei spiegeln die Sequenzierfragmente (Reads) das Verhältnis an maternaler zu fetaler cfDNA wider (90% : 10%, rot-graues Muster). Auf die Gesamtmenge bezogen stammen bei nicht trisomen Feten 1,5% aller NGS-Reads vom Chromosom 21; bei der Trisomie führt die zusätzliche Menge an fetaler cfDNA zu mehr NGS-Reads. Allerdings wirkt sich die Zunahme an NGS-Reads rein rechnerisch erst in der zweiten Nachkommastelle aus (1,5% versus 1,575%). Diese extrem geringe Differenz stellt hohe Anforderungen an die Analytik und muss durch statistische Signifikanztests abgesichert werden.
gMPS versus aMPS
Zwei Methoden für die nicht-invasive Pränataltestung (NIPT) stehen derzeit zur Auswahl: die gesamtgenomische massive Parallelsequenzierung (gMPS) und die Sequenzierung nach spezifischer Anreicherung von Zielsequenzen aus dem betroffenen Chromosom (aMPS). gMPS ist aufwendiger, dafür können aber alle verfügbaren cfDNA Fragmente aus dem mütterlichen Blut unabhängig von ihrer DNA-Sequenz quantitativ analysiert werden. Zudem kann bei diesem Ansatz eine unzureichende Messpräzision im unteren Nachweisbereich – wenn also beispielsweise in einer Blutprobe statt 10% nur 5% cffDNA vorliegen – dadurch kompensiert werden, dass man die Sequenziertiefe erhöht. Die Sequenziertiefe gibt an, wie oft ein bestimmter Abschnitt eines Genoms sequenziert wurde.
aMPS hingegen basiert auf der Anreicherung von Zielsequenzen auf den Chromosomen 13, 18, 21, X und Y mittels PCR oder Hybridisierungsverfahren. Dazu müssen mindestens zwei entsprechende Bindungsstellen für die Anreicherungsoligonukleotide im Testkit (PCR-Primer plus ggf. weitere Sonden) intakt vorliegen. Ist dies nicht der Fall, so erniedrigt sich die Anzahl an den verfügbaren, quantifizierbaren Markersequenzen, was die Test-spezifische Messpräzision erniedrigt. Diese Einschränkung ist abzuwägen gegen den Vorteil, dass die Amplikons in der Folge mit einer viel niedrigeren Sequenziertiefe analysiert werden können, was mit einer deutlichen Kostenreduktion einhergeht.
Fetale Fraktion
Die fetale Fraktion wird als die kritischste analytische Variable eines NIPT-Ergebnisses angesehen. Liegt hier ein hoher Anteil vor, so findet man auch mehr Reads mit entsprechend einfacherer Trisomie-Detektion. Dabei ist aber entscheidend, wie die fetale Fraktion gemessen wurde, denn nicht jede Methode liefert dafür präzise und zuverlässige Ergebnisse.
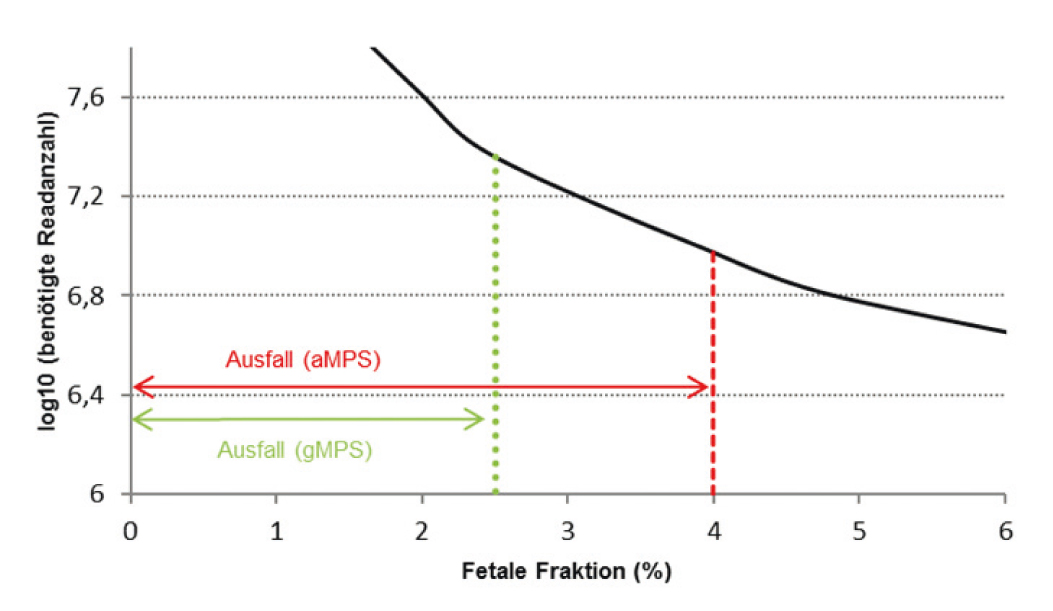
Auffällig ist vor allem, dass die meisten aMPS-Verfahren einen minimalen Grenzwert der fetalen Fraktion von 4% aufweisen. Dies kann man sich unter anderem damit erklären, dass durch die Anreicherungsmethode ein zusätzlicher systematischer Fehler eingeführt wird, der sich zum Fehler der eigentlichen Sequenzierung bzw. der Read-Quantifizierung addiert. Deshalb weisen gMPS-basierte NIPTs mit hoher Sequenziertiefe deutlich bessere Sensitivitäten bei gleichzeitig niedrigerem fetalem Fraktionsgrenzwert auf (z. B. 2%)[3]. Durch die tiefere Sequenzierung reduziert sich sowohl die Variation zwischen den Sequenzierläufen als auch innerhalb einer Sequenzierung, wodurch eine schärfere Abgrenzung von betroffener zu nicht betroffener Probe erfolgen kann.
Abb. 2 demonstriert den Einfluss der Sequenziertiefe grafisch. Sie zeigt, dass man die Sensitivität eines NIPT-Verfahrens auf jeden gewünschten Wert hochskalieren kann, wenn die Sequenziertiefe entsprechend angepasst wird. Gängige NGS-Methoden mit Anreicherung von Zielsequenzen (aMPS) haben einen minimalen Grenzwert der fetalen Fraktion von 4% etabliert. Proben mit einer fetalen Fraktion < 4% können also mit aMPS nicht analysiert werden. Mit gMPS-Methoden erreicht man dagegen durch Erhöhung der Reads eine Nachweisgrenze derzeit minimal 2%[3] (Stand 2016).
Dieser Unterschied ist durchaus bedeutsam, denn in einem durchschnittlichen Patientenkollektiv mit gemischter Risikokonstellation für eine Trisomie können bis zu 10% der Patientenproben eine fetale Fraktion von 2–4% aufweisen. Zusammenfassend ist die fetale Fraktion per se also eine wichtige Variable bezüglich der NIPT-Ergebnisvalidität, doch sie muss immer im Kontext der Messmethode und Sequenziertiefe des verwendeten MPS-Verfahrens begutachtet werden.
Leistungsdaten
2015 wurden im Rahmen einer Meta-Analyse von 37 Studien die diagnostischen Gütekriterien aller derzeit verfügbaren NIPT-Verfahren für die Trisomien 13, 18 und 21 sowie geschlechtschromosomale Aneuploidien zusammengefasst und gewichtet. Als Goldstandard diente dabei der invasiv bestimmte Karyotyp oder die postnatale, klinische Untersuchung. Ein Befund im engeren Sinne lag nicht vor[5].
Die Spezifität der NIPT-Verfahren war durchwegs hervorragend. Für die Trisomie 21 wurden weniger als 0,1% falsch-positive Befunde erfasst, und auch bei allen anderen oben genannten Konstellationen lag diese Rate unter 0,25%.
Auch die höchste Sensitivität fanden die Autoren für die Trisomie 21; hier wurden über 99% der positiven Fälle korrekt erkannt. Nicht ganz so gut schnitten die NIPT-Verfahren bei Trisomie 18 mit 96% richtig-positiven Befunden ab, und bei der Trisomie 13, den Trisomien XXX, XXY und XYY sowie der Monosomie X betrug die Sensitivität nur noch rund 90%.
Das bedeutet, dass ein positives Testergebnis mit an Sicherheit grenzender Wahrscheinlichkeit auch tatsächlich die gefundene Erkrankung anzeigt. Trotz der geringen Inzidenzen reicht der positiv prädiktive Wert der NIPT bei der Trisomie 21 nahe an die 100% heran und liegt auch bei den übrigen Erkrankungen in der Größenordnung von 90%.
Im Fall der Trisomie 21 ist auch ein negatives Testergebnis sehr zuverlässig, während bei den übrigen Konstellationen mit 4–9% falsch-negativen Resultaten gerechnet werden muss. Diese Unsicherheit lässt sich u. a. damit erklären, dass bei einer Trisomie 13 oder 18 im Mittel weniger fetale DNA ins mütterliche Blut gelangt als bei Trisomie 21[6], und dass die Basenzusammensetzung der entsprechenden Chromosomen einer signifikanten Variation unterliegt[7]. Die Beseitigung dieser Unsicherheit erfordert anspruchsvolle, bioinformatische Normalisierungsstrategien, welche jedoch das Auftreten falsch-negativer Ergebnissen nie vollständig verhindern können.
Speziell bei der Monosomie X (Karyotyp: 45,X0 statt 46,XX) und weiteren geschlechtschromosomalen Aneuploidien muss auch mit einem gehäuften Auftreten von feto-plazentaren, genetischen Mosaiken gerechnet werden, sodass eine Diskordanz zwischen fetalem und plazentarem Karyotyp auftreten kann[8]. So nimmt beispielsweise das Risiko für einen Verlust des X-Chromosoms im Rahmen einer Mosaikkonstellation im Karyotyp der Mutter mit dem Alter zu[9]. Auch zeigen ca. 60% aller Schwangerschaften mit einer fetalen Monosomie X ein plazentares Mosaik[10].
Zusammenfassend sind nicht-invasive Pränataltests auf der Basis von NGS vor allem bei Verdacht auf eine Trisomie 21 allen anderen nicht-invasiven Screening-Methoden überlegen. Bei einer Spezifität von nahezu 100% ist das Auftreten von falsch-positiven Ergebnissen sehr unwahrscheinlich. Da die positiv prädiktiven Werte – bedingt durch die extrem geringe Inzidenz – aber je nach Befund zwischen 85% und 95% schwanken können, empfehlen mehrere Fachgesellschaften dennoch, einen positiven NIPT-Befund mit invasiver Diagnostik zu verifizieren.
Die Sensitivitäten betragen dagegen teilweise „nur" etwa 90%. In den meisten anderen Bereichen der Labordiagnostik wäre dies ein sehr guter Wert, aber an eine vorgeburtliche Untersuchung mit gravierenden Konsequenzen für Eltern und Kind werden extreme Anforderungen gestellt. Man muss den Eltern also klar sagen, dass ein negativer NIPT-Befund das Vorliegen einer Aneuploidie sehr unwahrscheinlich macht, aber nie ganz ausschließt. Ein Restrisiko bleibt grundsätzlich bei jedem diagnostischen Test bestehen, und sei er noch so zuverlässig.

Dr. Thomas Harasim, Zentrum für
Humangenetik und Laboratoriumsdiagnostik
Dr. Klein, Dr. Rost und Kollegen
thomas.harasim[at]medizinische-genetik[dot]de

