Qualitätssicherung im molekularbiologischen Labor
Erfahrungsbericht
Die Validierung molekularbiologischer Tests ist noch immer eine Herausforderung; das gilt insbesondere für die quantitativen Real-Time-PCR-Verfahren.
Das Zentrum für Laboratoriumsmedizin, Mikrobiologie und Krankenhaushygiene an der Carl-Thiem-Klinikum gGmbH Cottbus wurde 1995 als Labor für Mikrobiologie, Virologie und Tuberkulose gegründet. Es hat dabei die rasante Entwicklung der Nukleinsäureamplifikations-Techniken von der einfachen Blockcycler-PCR mit Nachweis im Agarose-Gel bis zur Sonden-gestützten Real-Time-PCR und DNA-Sequenzierung durchlaufen. Mittlerweile sind die molekularbiologischen Labore der Mikrobiologie, Humangenetik und Molekularpathologie unter einem Dach vereint und teilen sich das hoch qualifizierte Personal und die Laborinfrastruktur. Seit 2010 sind Mikrobiologie und Humangenetik mit kommerziellen und Inhouse-PCR-Methoden nach DIN 15189 akkreditiert.
Der gesetzliche Rahmen der Qualitätssicherung
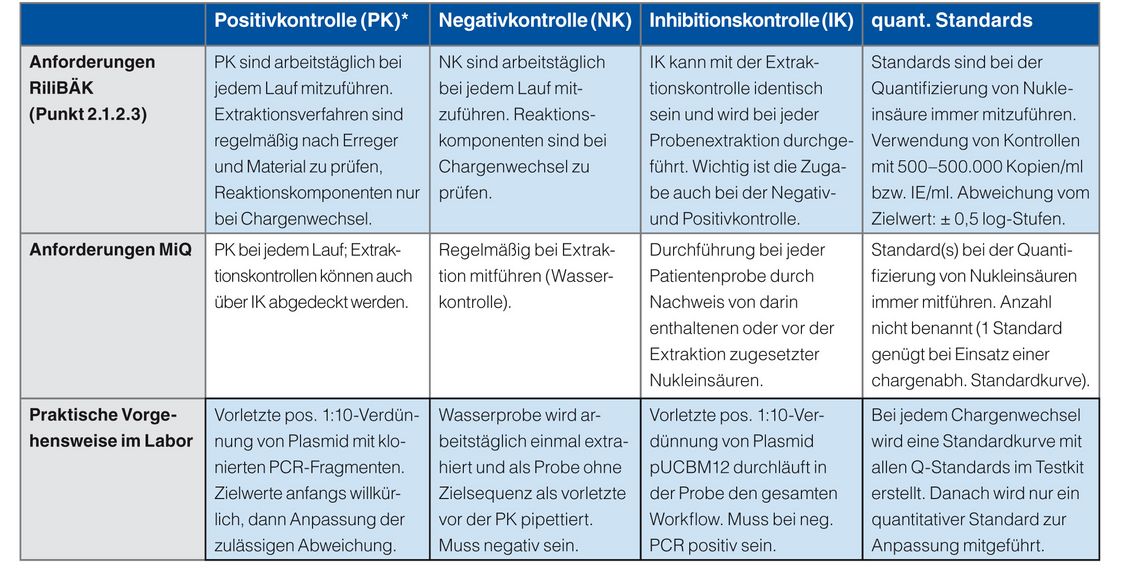
Schon früh erkannten Mikrobiologen und Virologen die hohe Bedeutung von Qualitätsstandards im PCR-Labor; bereits 2001 gab eine deutsche Gruppe von Molekular- und Mikrobiologen erstmals einen Leitfaden für die Qualitätssicherung molekularbiologischer Methoden heraus (MiQ 01: Nukleinsäure-Amplifikationstechniken: Qualitätsstandards in der mikrobiologischen Diagnostik, Hrs. Herrmann M. et al., 3. Auflage, Urban & Fischer 2011) . Mit der Neufassung der RiliBÄK im Jahr 2014 wurden daraus gesetzlich verbindliche Vorgaben für die molekularbiologischen Methoden in den Fachbereichen Humangenetik und Mikrobiologie/Virologie. Sie beinhalten u. a. den Einsatz der unten tabellarisch aufgelisteten Kontrollproben.
Praktische Umsetzung
Vier Arten von Kontrollen (positiv, negativ, Inhibition, quantitativ) sind beim Einsatz von PCR und Real-Time-PCR sinnvoll und werden arbeitstäglich für jede Methode mitgeführt. In kommerziellen Kits sind die Kontrollen enthalten, für unsere Inhouse-Verfahren stellen wir die Positivkontrollen aus unterschiedlichen Nukleinsäuren selbst her. Dazu gehören genomische DNA, virale RNA, Plasmide und kommerziell verfügbare Kontrollen mit Mengenangaben. Wir klonieren das nachzuweisende PCR-Produkt, stellen eine 1:10-Verdünnungsreihe her und führen erneut eine PCR durch. Die vorletzte Verdünnung, in der sich das PCR-Produkt noch nachweisen lässt, deklarieren wir als unsere Positivkontrolle.
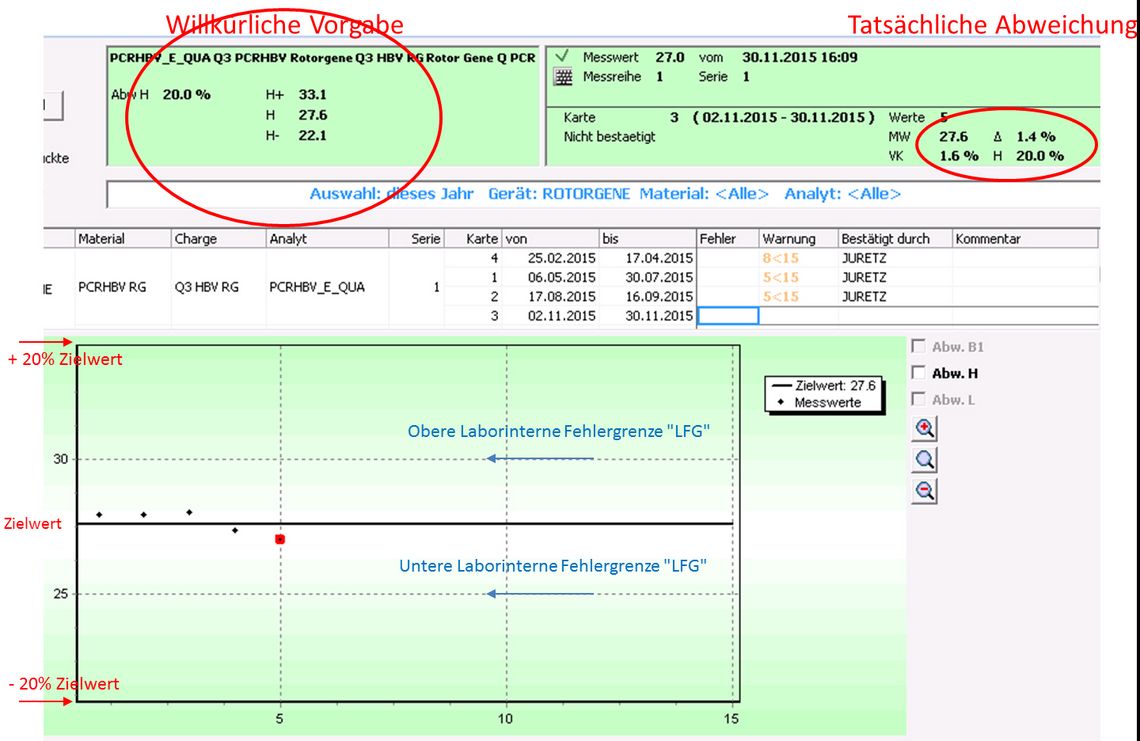
Zusätzlich nutzen wir den arbeitstäglich ermittelten ct-Wert der Positivkontrolle (Zielwert ermittelt durch drei Wiederholungsmessungen) als Marker für die Überwachung der PCR-Performance. Er gibt uns Auskunft über die Stabilität der Methode und den Einfluss der Reaktionskomponenten, z. B. bei Chargenwechsel.
Als Negativ-Kontrolle dient eine targetfreie Wasserprobe, die einmal arbeitstäglich den kompletten Workflow durchläuft.
Die Inhibitionskontrolle, mit der wir auch das Extraktionsverfahren überprüfen, ist die bedeutendste, denn sie überwacht den Einfluss der Patientenprobe auf die PCR. Sie muss positiv sein, um ein negatives Ergebnis bei einer Erreger-PCR zu bestätigen. Dazu dokumentieren wir intern den ct-Wert jeder Patientenprobe. Er wird zwar nicht im Befund ausgegeben, findet aber bei der technischen und medizinischen Validation Berücksichtigung.
Lässt sich für die Inhibitionskontrolle kein ct-Wert ermitteln, dann liegt eine Totalinhibition vor; partiell ist sie, wenn die Amplifikation verzögert ist. Liegt eine Inhibition vor, kann die Probe durch Fällung mit Ethanol weiter gereinigt werden. Bleibt die Probe inhibiert, wird das in einem Kommentar auf dem Befund dokumentiert und neues Material angefordert.
Die quantitativen Standards, also Kontrollproben mit bekannter Nukleinsäure- oder Erregerkonzentration, sind für jedes Routinelabor eine Herausforderung, weil Konzentrationsangaben oft nur Näherungswerte sind und die Mengeneinstellung deshalb nur grob oder gar nicht erfolgen kann. Zumal diese Standards auch noch international, z. B. mit WHO-Vorgaben, abgeglichen werden sollten.
Für unsere Viruslast-Bestimmungen setzen wir Kit-unabhängige, quantitative Standards, die Qiagen für den Rotor-Gene Q zur Verfügung stellt, als Laufkontrollen ein. Zusätzliche Kontrollen stammten von Minerva Biolabs und Vircell Microbiologists.
Die RiliBÄK-Vorgaben (Zielwert in Kopien/ml ± 0,5 log-Stufen) konnten wir sogar beim Chargenwechsel einhalten. Das gilt für den Nachweis von CMV- und HBV-DNA bzw. den Nachweis von HCV- und HIV-RNA bei Verwendung von Kontrollen mit 500–500.000 Kopien/ml bzw. IE/ml.
Zusammenfassung
Die Kontrollen in der Molekularbiologie sind wichtige Werkzeuge, um die verschiedenen Prozessparameter (Extraktion, Lagerung, Amplifikation) zu überwachen, Rückschlüsse auf Schwachstellen zu erhalten und dadurch die Qualität der Molekularbiologie zu sichern bzw. zu verbessern.
Die Überwachung der Extraktionsverfahren erfolgt bei uns aus jeder Patientenprobe, denn auch die beste PCR hilft nichts bei schlechter Extraktion. Der Einsatz von Laufkontrollen über die Chargengrenzen hinaus kann dazu führen, dass die Abweichungen von Charge zu Charge kleiner werden. Eine wichtige Information zur Funktionskontrolle „humane DNA“, die z. B. als Extraktionskontrolle eingesetzt wird, ist die Angabe der Anzahl der Genome. Ist diese bekannt, kann dadurch die Richtigkeit eines Ergebnisses (z. B negatives Ergebnis bei der HPV-PCR oder positiver Nachweis in der Molekularpathologie) weiter untermauert werden.
QIAGEN Instruments AG
Dr. Antje Plaschke-Schlütter
antje.plaschke-schluetter
@qiagen.com
www.qiagen.com
Autorenkontakt:
Dr. Th. Juretzek
Carl-Thiem-Klinikum Cottbus, Zentrum für Molekulare Diagnostik
+49 355/46-3091
t.juretzek[at]ctk[dot]de