Die Analyse von zirkulierender freier Tumor-DNA aus dem Blut stellt eine wertvolle Ergänzung zum Mutationsnachweis aus Tumorgewebe (Biopsie, Resektat) dar, und zwar insbesondere dann, wenn die Gewebeentnahme schwierig oder für den Patienten zu belastend ist. Als sog. „Liquid Biopsy“ gewinnt diese noch relativ junge Technik in der onkologischen Diagnostik zunehmend an Bedeutung – aktuell beispielsweise zum Nachweis der Resistenz-vermittelnden EGFR-p.T790M-Mutation bei Progression eines nicht-kleinzelligen Lungenkarzinoms (NSCLC) unter der Behandlung mit einem konventionellen EGFR-Tyrosinkinase-Inhibitor (EGFR-TKI) der ersten oder zweiten Generation. Der Nachweis dieser Mutation ist für die Verordnung des Drittgenerations-Tyrosinkinase-Inhibitors Osimertinib in der Zweitlinientherapie vorgeschrieben und wurde als erste und bisher einzige Liquid-Biopsy-Untersuchung in den EBM-Vergütungskatalog aufgenommen (Ziffer 19460).
In der vorliegenden Arbeit werden die einzelnen Schritte der Liquid Biopsy am Beispiel dieser EGFR-p.T790M-Mutation detailliert beschrieben und methodenkritisch bewertet. Ein besonderer Fokus liegt auf der korrekten Probengewinnung einschließlich Transport ins Labor (Präanalytik). Weitere Abschnitte befassen sich mit der Auswahl des geeignetsten Nachweisverfahrens von der klassischen qPCR bis zum Next Generation Sequencing inkl. Methodenvalidierung (Analytik) sowie der Gestaltung des Befundberichts (Postanalytik).
Die hier beispielhaft dargestellten Techniken sind auch auf andere Mutationen, Indikationen und Körperflüssigkeiten anwendbar. Es ist zu erwarten, dass die Liquid Biopsy in Zukunft nicht nur für Einzelanalysen, sondern auch für ganze Mutations-Profile eingesetzt wird.
Dieser Beitrag gibt die Ergebnisse eines Expertenworkshops wieder, der am 9. und 10. März 2018 in Frankfurt am Main stattfand.
Eine ausführliche Version ist in Buchform erschienen [1].
Schlüsselwörter: Liquid Biopsy, NSCLC, EGFR-p.T790M-Mutation, Osimertinib
In den letzten 20 Jahren wurden zahlreiche onkologische Treibermutationen identifiziert, die ursächlich für das Tumorwachstum verantwortlich sind. Ihr Nachweis hat die therapeutischen Möglichkeiten in der Onkologie revolutioniert, denn vielfach stellen die mutierten Genprodukte Zielstrukturen für neue Medikamente dar. So war der Nachweis, dass der Tyrosinkinase-Inhibitor Gefitinib nur gegen Tumoren mit aktivierenden Mutationen im Rezeptorgen für den epidermalen Wachstumsfaktor (EGFR) wirksam ist, der Beginn der personalisierten Therapie beim nicht-kleinzelligen Lungenkarzinom (NSCLC).
Trotz beeindruckender Erfolge dieser Behandlungsstrategie ist eine dauerhafte Heilung beim fortgeschrittenen NSCLC bislang allerdings noch nicht möglich, denn gegen die Wirkung solcher Inhibitoren entwickeln Krebszellen früher oder später Resistenzen. Sie entstehen durch neue Mutationen in den Tumorzellen oder durch Selektion von primär resistenten Subklonen. Um solch eine Resistenzentwicklung rechtzeitig zu erkennen und das Therapieschema gegebenenfalls anpassen zu können, sind molekularpathologische Kontrollen erforderlich.
Diese erfolgen idealerweise direkt aus dem Tumorgewebe (Resektat, Biopsie). Da die Entnahme aus der Lunge für den Patienten eine erhebliche Belastung darstellt, rückte als Alternative in den letzten Jahren die sogenannte „Liquid Biopsy“ immer mehr in den Vordergrund – also die molekulargenetische Untersuchung von Tumor-DNA im Blut (circulating tumor DNA, ctDNA).
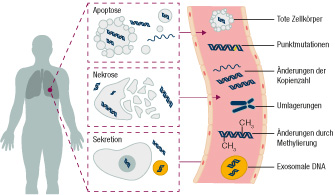
Basierend auf der Beobachtung, dass Körperzellen ganz allgemein und Tumorzellen im Speziellen Nukleinsäuren in die Blutbahn abgeben (siehe Abb. 1), können damit therapierelevante genomische Abschnitte analysiert werden, die sowohl aus dem Primärtumor als auch aus etwaigen Metastasen stammen.