Der rasante Fortschritt neuer Technologien der Hochdurchsatz-Sequenzierung in den letzten Jahren hat völlig neue Strategien zur umfassenden, kulturunabhängigen Charakterisierung von mikrobiellen Gemeinschaften eröffnet. Zahlreiche große internationale Studien haben Zusammenhänge unterschiedlichster Krankheitsbilder mit der Zusammensetzung der intestinalen Mikrobiota gezeigt. Die zugrunde liegenden pathophysiologischen Mechanismen und ursächlichen Zusammenhänge dieser mikrobiellen Wechselwirkungen sind jedoch nur in wenigen Beispielen bekannt und Ergebnis umfangreicher Forschung an gnotobiotischen oder keimfreien Tiermodellen sowie großer Interventionsstudien. Der Transfer dieser Erkenntnisse der biomedizinischen Grundlagenforschung in die klinische Anwendung ist sicherlich eine der größten aktuellen Herausforderungen im nach wie vor jungen Feld der Mikrobiomforschung. Das Gelingen dieses translationalen Übergangs hängt vom besseren Verständnis der großen interindividuellen Mikrobiomunterschiede sowie vom besseren Wissen über die unzähligen Einflussfaktoren ab, von der Ernährung, sportlicher Aktivität, oder dem Alter bis hin zur kaum überschaubaren und wechselseitigen Beeinflussung der Mikrobiota durch Medikamente oder Nahrungsergänzungsmittel.
Qualitätssicherung
Mikrobiom-modifizierende neue Therapien erfordern jedoch neben gut kontrollierten Kohortenstudien und wachsendem Erkenntnisgewinn über Wirts-MikrobiotaBeziehungen auch die sorgfältige Umsetzung qualitätskontrollierter und standardisierter Protokolle im Sequenzierlabor und bei der komplexen Datenanalyse sowie eine kontinuierliche Qualitätsüberwachung und Akkreditierung der Analytik. Darüber hinaus werden neue Werkzeuge zur Funktionsanalyse von Mikrobiomsignaturen benötigt. Nur wenn diese Voraussetzungen erfüllt sind, kann die Mikrobiom-Diagnostik zukünftig als wichtiges Instrument zur Prognose von Krankheitsverläufen sowie für die Entwicklung neuer individualisierter Therapien auf der Grundlage von Mikrobiomuntersuchungen erfolgreich angewandt werden.
Diese Voraussetzungen sind derzeit weitestgehend als noch nicht erfüllt anzusehen. Es fehlen harmonisierte Standardarbeitsanweisungen (SOPs) und Richtlinien zur standardisierten und qualitätskontrollierten Durchführung. Neben dem Fehlen von Goldstandards für die Analytik unterscheidet sich die in den einzelnen Laboren etablierte Methodik teils erheblich, und kommerzielle Produkte sind nur für einzelne Teilschritte verfügbar [1]. Obwohl die Anwendbarkeit der Mikrobiom-Diagnostik in der klinischen Praxis zumindest in ausgewählten Fällen gezeigt wurde, ist eine Implementierung in gesetzliche Qualitätssicherungssysteme selten. Die Akkreditierung von Laboren auf der Grundlage von DIN-Normen (z. B. DIN EN ISO 15189 für medizinische Diagnoselabore) und die Zusammenarbeit mit nationalen Akkreditierungsstellen werden mit dem wachsenden Erkenntnisgewinn über das diagnostische Potential der Mikrobiom-Analytik erforderlich. Ein wirksames und regelmäßig durch interne und externe Audits überprüftes Qualitätsmanagementsystem mit der Implementierung von Standardarbeitsanweisungen, einer umfassenden Qualifikation des Personals, Rückverfolgbarkeit aller damit verbundenen Schritte und die gut dokumentierte regelmäßige Überprüfung von Instrumenten, Reagenzien und Analysensystemen sollte dem vorrangigen Ziel der Verbesserung der Patientenversorgung untergeordnet werden. Für akkreditierte Labore ist die Teilnahme an externen Qualitätsbewertungsprogrammen und die Prüfung von biologischen Referenzmaterialien obligatorisch. Insbesondere für das ebenso komplexe wie diverse Feld der Mikrobiom-Analytik stellt dies ein wichtiges Instrument dar, um einen Prozess der kontinuierlichen methodischen Verbesserung zu initiieren, der darauf abzielt, Fehler zu reduzieren und zukünftig valide diagnostische Befunde zu erstellen.
Erster Ringversuch
Der erste in Deutschland durchgeführte Inter-Laborvergleich fand 2014 statt [2]. An diesem Ringversuch, der im Zuge des DFG-geförderten Schwerpunktprogramms „SPP 1656: Intestinale Mikrobiota“ durchgeführt wurde, nahmen insgesamt neun deutsche und österreichische Zentren teil. Alle Teilnehmer erhielten ein Aliquot einer zuvor homogenisierten und mit zuvor quantifizierten, exogenen „Spike-Bakterien“ versetzten Stuhlprobe und die daraus extrahierte Nukleinsäurepräparation. Die insgesamt neun Teilnehmer aus dem deutschsprachigen Raum wandten die in ihrem Labor etablierte Methodik zur Analyse bakterieller 16S-rRNA-Gene mittels Hochdurchsatz-Sequenzierung an, wobei die Probe in den Laboren mehrfach den Analyse-Prozess durchlief. Ziel war die Erfassung der Abweichungen aller methodischen Schritte der etablierten komplexen Mikrobiom-Analytik, angefangen von der DNA-Extraktion über die Next-Generation-Sequencing-(NGS-)Verfahren bis hin zur bioinformatischen Spezies-Zuordnung und Erstellung/Übermittlung der Analyse-Reports. Die gute Nachricht: Alle Teilnehmer lieferten reproduzierbare Ergebnisse bei mehrmaliger Analyse derselben Stuhlprobe. Die Mikrobiom-Profile der einzelnen Labore waren jedoch kaum mit der ermittelten Zusammensetzung der jeweils anderen Labore vergleichbar. Der wichtigste Grund für diese Unterschiede lag in der Auswahl der Primer zur Amplifikation der 16S-rRN-Gene. Diese beobachtete, erschreckend hohe Inter-Labor-Varianz wurde mittlerweile auch in zahlreichen weiteren Studien internationaler Konsortien wie dem Microbiome Quality Control (MBQC) project [3] oder dem Microbiome Standards Project [4] beobachtet und berichtet.
Jährliche Ringversuche
Aus den Erkenntnissen dieser Analysen entstand die Überlegung einer jährlichen Durchführung von Ringversuchen für die Mikrobiom-Analytik. In Zusammenarbeit mit INSTAND Gesellschaft zur Förderung der Qualitätssicherung in medizinischen Laboratorien e. V. wurde deshalb ein jährlich stattfindender Ringversuch (Ringversuchsgruppe 580: Molekulargenetische Analyse des humanen Mikrobioms) ins Leben gerufen.
Der Ringversuch zur Mikrobiom-Analytik wurde unter der Leitung von Prof. Dr. Dr. André Gessner und Prof. Dr. Udo Reischl erstmalig 2015, in diesem Jahr also nun bereits zum fünften Mal mit jährlich steigenden Teilnehmerzahlen (aktuell: 19 Labore mit weltweitem Einzugsgebiet) durchgeführt.
Exemplarisch sollen hier die Konzeption und die Ergebnisse der Ringversuchsrunde des Jahres 2018 vorgestellt werden, die repräsentativ für die Ergebnisse der vorherigen Jahre stehen. Aufgrund der Fülle an Daten kann hieraus jedoch nur ein kurzer Ausschnitt näher erläutert werden. Als Konsequenz aus der kontinuierlich starken Varianz der von den Teilnehmern jährlich gemeldeten Ergebnisse seit Einführung des Ringversuchs, wurde hier ein verstärktes Augenmerk auf überprüfbare Anteile von definierten Bakterienspezies gelegt. Humane Stuhlproben eines Probanden wurden mit quantitativen Mengen von bakteriellen Zellen bekannter Spezies versetzt. Deren Anteil in den Stuhlproben war durch vorherige Zellzählung und anschließende Überprüfung der Nukleinsäure-Extrakte mittels Spezies-spezifischer quantitativer real-time PCR-Protokolle bekannt [5]. Diese zugesetzten exogenen Bakterien-Spezies sind nicht Bestandteil der „normalen“ humanen Mikrobiota, gehören unterschiedlichen phylogenetischen Gruppen an und besitzen darüber hinaus jeweils unterschiedliche Eigenschaften (Beschaffenheit der Zellwand, GC-Gehalt des Genoms, 16S-rRNA-Gen-Kopienzahl im Genom, taxonomische Differenzierbarkeit). Diese Diversität an molekularbiologischen und phänotypischen Eigenschaften wurde gewählt, da in der Theorie daraus eine größere Varianz in den nachfolgenden Analysen resultieren sollte. Die Bakterienspezies können mithilfe von spezifischen real-time PCR-Protokollen in der Probe quantifiziert werden. Zur Prüfung der DNA-Extraktionsverfahren wurde außerdem ein Aliquot der aus dieser Probe gewonnenen Nukleinsäurepräparation verschickt.
Da kein Goldstandard für die Mikrobiom-Analytik existiert, besteht eine humane Stuhlprobe aus einer Vielzahl von Unbekannten. Aus diesem Grund wurden zusätzliche Ringversuchsproben hergestellt, die ausschließlich aus einer definierten Mischung (relative Häufigkeiten zwischen 0,1 und 48,5 %) von 25 unterschiedlichen, kultivierten Bakterienspezies (eine „Mock Community“) bestand. Die Spezieszuordnung wurde zuvor durch Sanger-Sequenzierung ihrer kompletten 16S-ribosomalen Gene (V1–V9 Region) validiert und die 16S-rRNA-enkopien der Extrakte mithilfe eines pan-bakteriellen real-time PCR-Protokolls quantifiziert. Auf dieser Basis wurden die Extrakte genomischer DNA in bekannten Verhältnissen gemischt und anschließend für alle Ringversuchsteilnehmer aliquotiert. Die Pakete mit den Ringversuchsproben wurden anschließend auf Trockeneis zu den einzelnen Teilnehmern geschickt.
Erfolgskriterien
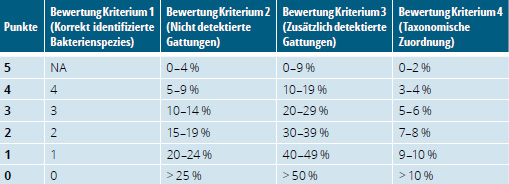
Folgende Kriterien für eine erfolgreiche Ringversuchsteilnahme wurden festgelegt und mithilfe eines Punktesystems bewertet (Tab. 1):