Das CRISPR/Cas9-System wurde in Bakterien entdeckt und als Technologie zur genetischen Modifikation in eukaryotischen Zellen adaptiert. Dies stellt einen Durchbruch in der biomedizinischen Forschung dar, da nun in kürzester Zeit und mit einfacher Methodik genetisch veränderte Zelllinien, primäre Zellen und ganze Organismen generiert werden können. Auch in der Klinik findet die CRISPR/Cas9-Technologie bereits ihren Nutzen. Hier diskutieren wir die Anwendung des CRISPR/Cas9-Systems zur genetischen Manipulation von hämatopoetischen Zellen und Generierung von Mausmodellen. Weiterhin behandeln wir die Verwendung der CRISPR/Cas9-Technologie als Screening-Werkzeug zur Identifizierung von Genen, die für verschiedene biologische Prozesse verantwortlich sind. Abschließend besprechen wir erste klinische Studien, die CRISPR/Cas9 in einem Immuntherapie-Ansatz verwenden, und zeigen potenzielle zukünftige Applikationen auf.
Schlüsselwörter: CRISPR/Cas9-Technologie, genetische Manipulierung, klinische Anwendung
Einleitung
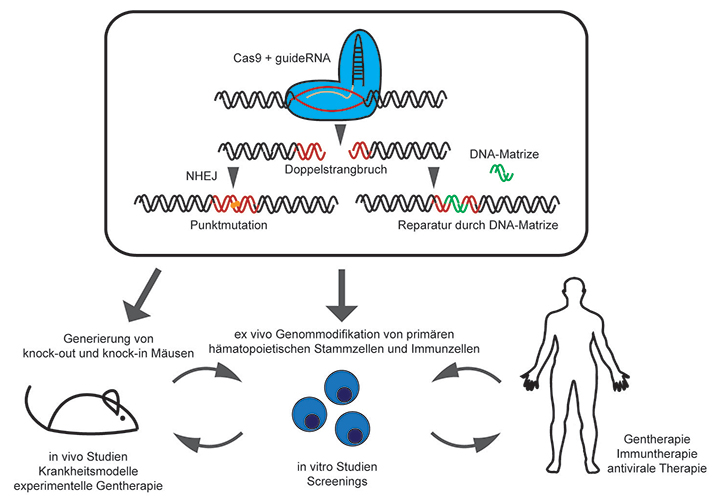
Die genetische Manipulation von Zellen und ganzen Organismen ist ein wichtiger Schritt bei der Entschlüsselung der Funktionen einzelner Gene oder Genprodukte bei immunologischen und anderen biologischen Prozessen. Während die Überexpression seit langer Zeit anwendbar ist, stellt die Inhibition der Genexpression eine größere Hürde dar. Verschiedene Techniken wie Zinkfinger-Nukleasen (ZFN) oder Transkriptionsaktivator-ähnliche Nukleasen (TALEN) wurden in den letzten Jahrzehnten benutzt, haben aber aufgrund von geringer Effizienz bzw. hohem Aufwand nicht zu einer routinemäßigen Anwendung geführt. Seit der Identifizierung des CRISPR/Cas9-Systems als Teil des adaptiven Immunsystems von Bakterien und Archaeen zur Abwehr von Phagen [1], ist die Technologie zur State-of-the-art-Methode für die genetische Veränderung eukaryotischer Zellen und Organismen avanciert [2]. Das System macht sich dabei die Endonuklease-Aktivität von Cas9 sowie zelluläre DNA-Reparatur-Mechanismen zunutze. Cas9 wird durch sequenzspezifische guide RNAs an eine bestimmte Stelle im Genom gelenkt und induziert DNA-Doppelstrangbrüche in diesen Bereichen. Diese DNA-Doppelstrangbrüche werden vom zelleigenen DNA-Reparatur-System durch nicht-homologe Verbindung von offenen Enden (non-homologous end-joining, NHEJ) repariert, wobei zufällige Insertions- oder Deletions-Mutationen eingeführt werden können, die in den meisten Fällen zur funktionalen Inaktivierung des betreffenden Gens führen [3–5]. Durch Modifizierung des Cas9-Enzyms kann die Technik außerdem zur zielgerichteten Aktivierung oder Unterdrückung der endogenen Genexpression oder sogar zur Markierung spezifischer genomischer Regionen genutzt werden [6, 7].
Manipulation von hämatopoetischen Zellen
Ein detailliertes Verständnis der molekularen Signalwege, die Immunantworten zugrunde liegen, erfordert die systematische Genmanipulation in den beteiligten Zellen. Seit der Adaptation des bakteriellen CRISPR/Cas9-Systems zur genetischen Modifizierung von eukaryotischen Zellen [4, 5] findet die Technik in vielen wissenschaftlichen Bereichen Anwendung.
Die hohe Effizienz und Präzision der CRISPR/Cas9-Technologie ermöglicht neben der Modifikation von immortalisierten Zelllinien, die theoretisch für unbegrenzte Zeit kultiviert werden können, auch die Behandlung von primären hämatopoetischen Stammzellen und Immunzellen, die eine begrenzte Lebensdauer haben und bei denen zeitintensive Selektions-Prozesse unmöglich sind [8, 9]. Dabei erweitern Cas9-transgene Mäuse die Anwendbarkeit der CRISPR/Cas9-Technologie, die derzeit noch mit Schwierigkeiten bei der Einführung der einzelnen CRISPR-Komponenten in einige Zelltypen verbundenen ist [8]. Induzierbare Systeme erlauben zudem die zeitlich kontrollierte Modifikation und Deletion von beispielsweise letalen Genen [10].
Die CRISPR/Cas9-vermittelte Manipulation von hämatopoetischen Vorläufer-Zellen und der Transfer dieser Zellen in (sub-)letal bestrahlte Mäuse stellt eine schnelle Methode zur Generierung von Mäusen mit Mutationen im hämatopoetischen Kompartiment dar; eine Methode, die – nicht zuletzt durch die Möglichkeit, mehrere Gene gleichzeitig zu mutieren – sowohl in der Immunologie als auch in der Krebsforschung von großer Bedeutung werden könnte [10, 11]. Da die CRISPR/Cas9-vermittelte Manipulation humaner hämatopoetischer Vorläufer-Zellen deren Differenzierungs-Potenzial nicht beeinträchtigt [12], könnte sie außerdem die Grundlage für neue Zell-basierte Therapie-Ansätze bilden.
Nagermodelle
Die Generierung von Knock-out-Mäusen durch konventionelle Methoden ist arbeits- und zeitintensiv. Genetische Veränderungen von embryonalen Stammzellen (ES-Zellen) durch homologe Rekombination, Zucht von chimären Mäusen mit Keimbahn-integrierten Mutationen und Kreuzungen heterozygoter Mäuse zur Generierung homozygoter Nachkommen können mehrere Monate bis Jahre einnehmen. Die Erzeugung von Mäusen mit Mutationen in mehreren Genen erfordert die Kreuzung verschiedener Mauslinien mit Veränderung in einzelnen Genen. Die Anwendung der CRISPR/Cas9-Technologie zur Generierung von Mausmodellen ist revolutionär. Mutationen können mit hoher Effizienz meist in beiden Kopien eines Gens und sogar in mehreren Genen gleichzeitig eingeführt werden. Dies wird durch Transfektion von ES-Zellen oder Injektion von Zygoten mit Cas9-mRNA oder -Protein zusammen mit einer sequenzspezifischen guide RNA erreicht. Neben zufälligen Deletionen und Insertionen können durch die Zugabe von DNA-Matrizen auch gezielte Punktmutationen und sogar loxP-Stellen oder Reporter-Gene ins Genom eingeführt werden [13–15]. Diese Errungenschaften verringern nicht nur die Zeit, sondern auch die Kosten, die notwendig sind, um ein passendes Mausmodell zu generieren [13]. Da zur Generierung von genetisch veränderten Tieren mittels CRISPR/Cas9 keine ES-Zellen mehr notwendig sind, ist es nun auch möglich, vorklinische Modelle von anderen Spezies herzustellen. Interessant ist hierbei vor allem die Generierung von genetisch modifizierten Ratten [16], welches als eines der besten Modellsysteme in der Multiple-Sklerose-Forschung zählt. Eine immer wiederkehrende Frage ist, wie spezifisch das CRISPR/Cas9-System ist und wie viele ungewollte Mutationen in anderen genomischen Regionen eingeführt werden. Wenngleich eine endgültige Antwort auf diese Frage im Moment nicht gegeben werden kann, scheint das CRISPR/Cas9-System verglichen mit anderen genommodifizierenden Methoden recht spezifisch zu sein [13] (eigene Beobachtung). Um CRISPR/Cas9-vermittelte Genom-Modifikationen aber noch spezifischer zu machen, wurden vor Kurzem spezifischere Cas9- bzw. guide RNA-Varianten entwickelt [17, 18]. Alles in allem stellt die CRISPR/Cas9-Technologie eine effiziente und einfache Methode zur schnellen Generierung genetischer Modifikationen in Mäusen und anderen Nagern dar, ein Fortschritt, der vor allem die immunologische Forschung, die sehr stark von Mausmodellen profitiert, immens beschleunigen wird.
Die Verwendung von CRISPR/Cas9 als Screening-Methode
Gene, die an bestimmten biologischen Prozessen beteiligt sind, können durch systematische genomweite Screenings identifiziert werden. Bisher wurden solche Screenings in eukaryotischen Zellen mithilfe von RNA-Interferenz (RNAi) durchgeführt. Diese Methode hat jedoch den Nachteil, dass sie die Expression von Genprodukten nur partiell unterdrückt und nicht komplett verhindert. Außerdem sind Off-target-Effekte keine Seltenheit, sodass ein RNAi-basiertes Screening-Experiment viele falsch positive oder falsch negative Ergebnisse hervorbringen kann. Verschiedene Studien haben gezeigt, dass CRISPR/Cas9-basierte Screenings mithilfe von genomweiten guide RNA-Bibliotheken in Zelllinien durch hohe Spezifität und Effizienz zuverlässig zur Identifizierung von Genen, die für bestimmte biologische Prozesse essenziell sind, führen können [19, 20] (guide RNA-Bibliothek: Eine Mischung aus mehreren tausend guide RNAs, die zusammen für alle Gene eines Genoms Sequenzspezifizität zeigen und damit die Mutation aller dieser Gene durch die Rekrutierung und Aktivierung von Cas9 hervorrufen können). Insbesondere kann diese Technologie dazu beitragen, komplexe immunologische Prozesse in primären Zellen besser zu verstehen.
In einer Studie von Parnas et al. [9] wurden aus dem Knochenmark Cas9-transgener Mäuse primäre dendritische Zellen gewonnen und mit einer lentiviralen genomweiten guide RNA-Bibliothek infiziert. Die transduzierten Zellen wurden mit LPS stimuliert und die Produktion des pro-inflammatorischen Zytokins TNFα gemessen. Zellen, die entweder mit verstärkter oder verringerter TNFα-Produktion auf LPS reagierten, wurden basierend auf der intrazellulären Färbung von TNFα mithilfe eines Durchflusszytometers sortiert und die guide RNAs sequenziert. Zum einen konnte dadurch die Methode validiert werden, da Gene gefunden wurden, deren Rolle in diesem Prozess fest etabliert ist. Zum anderen identifizierte die Studie bisher unbekannte Faktoren, die wichtig für die Expression oder Regulation von TNFα sind [9].
Auch kleine CRISPR/Cas9-basierte Screenings, bei denen Kandidaten-Gene zielgerichtet mutiert werden, sind in primären Immunzellen Cas9-transgener Mäuse möglich. Das zeigt eine Studie von Chu et al. [8], in der die Identifizierung von Genen, welche das Überleben, die Proliferation und die Plasmazell-Differenzierung von B-Zellen steuern, demonstriert wird. Die Autoren sagen voraus, dass solche Screenings auch in anderen primären Immunzellen möglich sind [8].
Hochinteressanterweise wurde kürzlich in einer Publikation von Jaitin et al. CRISPR/Cas9-basiertes Screening mit Einzelzell-RNA-Sequenzierung (CRISP-seq) in vitro und in vivo kombiniert [21]. Diese Experimente legen den Grundstein für die Analyse komplexer immunologischer Netzwerke. In der Studie behandelten die Autoren Knochenmarkzellen von Cas9-transgenen Mäusen mit einer Kombination aus Transkriptionsfaktor-spezifischen guide RNAs, die die Differenzierung von myeloiden Zellen beeinflussen. Diese guide RNAs waren mit Fluoreszenz- und genetischen Markern versehen, sodass einzelne Knock-out-Zellen identifiziert werden konnten. Durch die auf RNA-Sequenzierung basierende phänotypische Analyse und gleichzeitige Korrelation mit den verschiedenen guide RNAs konnten in den individuellen Knock-out-Zellen verschiedene Signalwege identifiziert werden. Diese Experimente waren nicht nur in der Lage, schon bekannte Signalkaskaden aufzudecken, sondern führten auch zur Aufklärung Zelltyp-spezifischer Funktionen einzelner Transkriptionsfaktoren, die die myeloide Differenzierung und Funktion nach Stimulation mit Zyokinen in vitro und in vivo diktieren [21].
Klinische Studien und potenzielle therapeutische Anwendungen
Therapien, bei denen hämatopoetische Stammzellen genetisch durch Überexpression einer funktionalen Kopie des im Patientengenom defekten Gens verändert werden, finden in der Klinik bereits Anwendung [22]. Da die Überexpression durch zufällige Integration der Transgene ins Genom mit Risiken wie Insertions-Mutagenese oder dysregulierter Expression verbunden ist, kann es unter anderem zur Abstoßung der modifizierten Zellen oder aber zur Aktivierung von Onkogenen und somit zur Entstehung von Krebs kommen [23]. Diese Risiken können durch transiente Expression oder sequenz-spezifische Veränderung des mutierten Gens minimiert werden.
Ein Beispiel für einen Gentherapie-Ansatz, der auf sequenzspezifischer Reparatur eines defekten Gens beruht, stellt eine Studie von Genovese et al. dar. In dieser Studie wurde der Gendefekt, der zur X-linked Severe Combined Immunodeficency führt, ex vivo in hämatopoetischen Stammzellen mithilfe von Genom-modifizierenden Zinkfingernukleasen (ZFNs) korrigiert [24]. Auch mithilfe der CRISPR/Cas9-Technologie lassen sich humane hämatopoetische Zellen sequenzspezifisch verändern [12]. Die geringere Komplexität des CRISPR/Cas9-Systems im Vergleich zu ZFNs und TALENs machen individuelle Therapien wesentlich einfacher.
Des Weiteren kann die CRISPR/Cas9-Technologie zur Deletion von Genen genutzt werden und könnte so schon bald Einzug in die Klinik erlangen. Tatsächlich ist die erste klinische Studie, die mithilfe der CRISPR/Cas9-Technologie veränderte Zellen zur Immuntherapie verwendet, bereits angelaufen [25]: Um das Immunsystem aggressiver gegenüber Krebszellen zu machen, wurde in T-Zellen von Krebs-Patienten das Gen für PD-1 ausgeschaltet und die modifizierten Zellen in die Patienten zurücktransferiert. Bisher zeigen Immuntherapien, bei denen PD-1 mit blockierenden Antikörpern inhibiert wird, hohen therapeutischen Erfolg, was eine Grundlage für diesen Ansatz darstellt. Kritiker hingegen sagen, dass die erfolgreiche Antikörper-basierte Therapie eine Gen-Therapie unnötig macht [25]. Eine weitere klinische Studie, bei denen T-Zellen nicht nur bezüglich der PD-1 Expression modifiziert, sondern zusätzlich mit Tumor-spezifischen T-Zell-Rezeptoren ausgestattet werden sollen, ist für die nahe Zukunft geplant.