Die Vielfalt der CDx-Methoden
Produktübersicht Companion Diagnostics
Die Zahl der molekularpathologischen Assays, die vor der Verordnung einer zielgerichteten Krebstherapie zwingend vorgeschrieben sind, hat sich in den letzten drei Jahren fast verdoppelt. Sie lassen sich in DNA-, mRNA- und Proteinassays gliedern. Neben einigen seit Langem eingeführten Verfahren wie etwa PCR, Immunhistochemie und Durchflusszytometrie halten zunehmend auch Hochdurchsatzverfahren für die Sequenzierung Einzug ins Routinelabor.
Bitte klicken Sie auf die nebenstehenden Firmenlogos, um zu den Produktübersichten zu gelangen.
DNA makes RNA and RNA makes protein. So lautete das zentrale Dogma der Molekularbiologie, das Francis Crick 1958 prägte, nachdem die Struktur der DNA aufgeklärt war. Auch wenn dieser Satz aufgrund neuerer Erkenntnisse, zum Beispiel über Retroviren, relativiert werden muss, ist er als roter Faden für eine molekularpathologische Methodenübersicht sehr gut geeignet.
Die Molekularpathologie untersucht heute vor allem die Zielmoleküle für die individualisierte Krebstherapie. Sie bedeutet für den einzelnen Patienten oft einen kleinen, für die Menschheit aber einen großen Schritt in Richtung eines verlängerten Lebens mit dem Tumor.
Die wachsende Bedeutung des Fachgebiets erkennt man an der Zahl der Tests, die unter dem Stichwort Companion Diagnostics (CDx) verpflichtend vorgeschrieben sind, um derartige Therapien einsetzen zu dürfen: Lag sie in unserer letzten Übersicht (Herbst 2013) noch bei 25, so sind es heute bereits 45.
PCR und Sequenzierung
Die Vielfalt der Tests lässt sich in drei Gruppen einteilen (s. Abb.): Die meisten betreffen DNA-Varianten, gefolgt von Expressionstests, unter denen wiederum die Proteinassays überwiegen. Führende Verfahren sind dank einfacher Durchführbarkeit die PCR (als Einzel- oder Multiplexansatz) und die Fluoreszenz-in-situ-Hybridisierung. Für diese Tests stehen zahlreiche zertifizierte Kits mit allen erforderlichen Reagenzien, Verbrauchsmaterialien und Kontrollen zur Verfügung (S. 198–199).
Als Besonderheit sei die „digitale PCR“ genannt, die die Vorteile von Emulsions-PCR, Magnetpartikeln und Durchflusszytometrie kombiniert (S. 200). Sie wird vor allem für die Analyse freier Tumor-DNA im Blut, die sog. Liquid Biopsy, eingesetzt.
Je größer die mutierten Gene sind (z. B. BRCA) bzw. je komplexer der Mutationsstatus eines Tumors im Krankheitsverlauf wird (beispielsweise beim Bronchialkarzinom), desto eher wird man sich anstelle der PCR für ein Hochdurchsatzverfahren entscheiden. Zurzeit hält das Next Generation Sequencing (NGS) mit gebrauchsfertigen Kits und leistungsfähigen Bioinformatiktools Einzug ins Routinelabor (S. 198 und 205). Hochparallele und relativ kostengünstige Analysen von mehreren hundert Mutationen pro Lauf sind auch mit der gut etablierten MALDI-TOF-Massenspektrometrie möglich (S. 198).
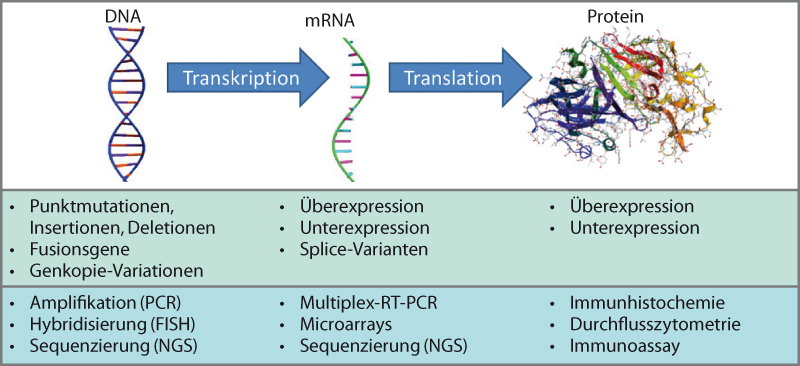
Expressionsassays
Die mRNA-Analytik wird vorwiegend mit Microarrays (Biochips), in neuerer Zeit auch mit NGS durchgeführt (S. 194 und 202). Wichtigstes Einsatzgebiet ist aktuell die Vorhersage des Therapieansprechens beim Mammakarzinom.
Als letztes Glied in der Ereigniskette des „zentralen Dogmas“ wäre die Expressionsanalytik auf Proteinebene zu nennen, die von der Historie der Methodenentwicklung her allerdings an erster Stelle steht. Bereits in den 1970er-Jahren wurde der Hormonrezeptorstatus beim Mammakarzinom mittells Immunhistochemie bestimmt, und dieses Verfahren gilt auch heute noch als Goldstandard für die Analyse solider Tumoren – mit entsprechend ausgereiften Kits und automatisierten Geräten (z. B. HER2-Status S. 199). In der Leukämiediagnostik ist dieser Goldstandard die Durchflusszytometrie (S. 199 und 201).
Dr. Gabriele Egert
Mitglied der Redaktion








