Respiratorische Erreger
Evaluation eines Multiplex-PCR-Assays
Das RespiFinder®-Panel von QIAGEN differenziert respiratorische Viren und Bakterien schnell und sicher. Dieses Multiplex-PCR-Verfahren vereinfacht den Arbeitsablauf erheblich und liefert im Vergleich zu Einzel-PCRs klinisch wertvolle Zusatzinformationen.
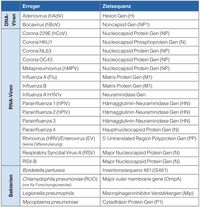
Als Auslöser für Atemwegsinfektionen kommen unterschiedlichste Viren sowie einige Bakterienstämme infrage, wobei die Symptome nur wenig zu deren Differenzierung und zur sicheren Diagnose beitragen. Mit einer Multiplex-PCR können die verschiedenen Erreger jedoch rasch differenziert werden. Das RespiFinder-Panel von QIAGEN unterscheidet 18 Viren und 4 Bakterienstämme.
Workflow
Nach der Nukleinsäure-Extraktion mit dem QIAamp® MinElute® Virus Spin Kit (empfohlene Prozedur) erfolgt eine Präamplifikation, die auch einen Reverse-Transkriptase-Schritt für die RNA-Viren beinhaltet. Daran schließt sich eine Hybridisierung mit spezifischen Primern gegen typische Nukleinsäuresequenzen der 22 nachzuweisenden Erreger an, gefolgt von einer Ligationsreaktion der hybridisierten Sequenzen. Die präamplifizierten Nukleinsäuren werden dann auf zwei Gefäße für die eigentliche Realtime-PCR aufgeteilt. Diese läuft im Rotor-Gene® Q mit fluoreszenzmarkierten Primern ab, wodurch die Reaktion sichtbar und die Schmelzkurvenanalyse überhaupt erst ermöglicht wird. Bis zu 13 verschiedene Zielnukleinsäuren können mit diesem Verfahren in einem einzigen Reaktionsansatz erfasst werden.
Methodenvergleich
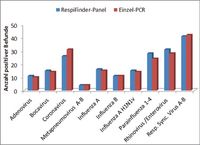
An insgesamt 165 klinischen Proben wurden in einer retrospektiven Studie die Ergebnisse für das RespiFinder-Panel mit den via LDT-PCR (Lab-Developed Test) erzielten Befunden verglichen[1]. Mit dem RespiFinder-Panel waren 157/165 (95%) der Proben für mindestens ein respiratorisches Pathogen positiv und 8/165 (5%) negativ. Mit den LDT-PCRs fielen 156/165 (94%) für mindestens ein respiratorisches Pathogen positiv und 9/165 (6%) negativ aus.
Die Auswertung über alle Proben und Pathogene zeigte in 143 Fällen (87%) völlig übereinstimmende Resultate. Bei den nicht zu 100% übereinstimmenden Pathogene wurden mit dem RespiFinder-Panel zusätzliche Erreger und damit Mischinfektionen nachgewiesen. Darunter fanden sich zum Beispiel in jeweils einer Probe hBoV, hCoV OC43, HRV/EV, FluA H1N1, C. pneumoniae und B. pertussis. Mit dem RespiFinder-Panel einzig nicht erfasst wurden 5 Fälle mit Coronaviren sowie eine schwache Mischinfektion mit RSV.
Zusammenfassend besagt diese Studie, dass man mit dem RespiFinder-Panel in einem einzigen Analysengang eine sehr große Anzahl von Erregern – einschließlich der häufigsten bakteriellen respiratorischen Keime – erfassen kann.
Saisonale Trends
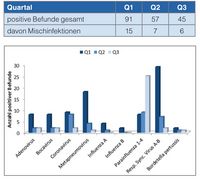
Am Uniklinikum Münster befindet sich das RespiFinder-Panel im Routineeinsatz. Hier wurde in einer weiteren Studie der saisonale Verlauf respiratorischer Infektionen geprüft. Die nebenstehende Tabelle gibt einen Überblick über die Daten der ersten drei Quartale 2014. Sie zeigt einen typischen jahreszeitlichen Trend mit einer Häufung positiver Befunde im Winterquartal und einem Rückgang auf etwa die Hälfte im Sommer.
Mischinfektionen mit bis zu drei viralen sowie einem viralen und einem bakteriellen Erreger lassen sich mit dem RespiFinder-Panel ebenfalls sehr gut nachweisen. Ihr Anteil lag im ersten Quartal bei 16%, im zweiten und dritten Quartal mit 12 bis 13% nur geringfügig niedriger.
In Abb. 2 ist dieses Ergebnis nach einzelnen Erregern aufgeschlüsselt. Der fallende Gesamttrend der Tabelle bestätigt sich auch bei jedem einzelnen Keim mit Ausnahme der Parainfluenza-Viren 1–4, die einen (typischen) gegenläufigen Verlauf zeigen.
Fazit
Der Multiplex-Nachweis mit dem RespiFinder-Panel von QIAGEN ist für die Routine-Diagnostik geeignet. Dieses Panel erkennt klinisch relevante respiratorische Erreger sicher und schnell und unterstützt so den behandelnden Arzt bei der Befundung und optimalen Therapieentscheidung. Dies betrifft nicht nur die Gabe von Antibiotika, sondern auch die Entscheidung über eine stationäre Aufnahme bzw. die Entlassung von Patienten.
Haftungsausschluss
Stand Januar 2015: erschienen in Trillium Diagnostik 2014, Band 12, Heft 4. Alle Inhalte und Kontaktdaten basieren auf Herstellerangaben zum Zeitpunkt der Veröffentlichung der Printausgabe. Die Trillium GmbH übernimmt keinerlei Haftung für die Aktualität und Richtigkeit der Firmenangaben. Bitte wenden Sie sich im Zweifel direkt an die Firmenzentrale des Herstellers. Fehler können Sie über unser Kontaktformular melden.
Quellenhinweis
Studiendaten stammen vom Poliklinikum San Matteo, Pavia, Italien, Routineergebnisse vom Universitätsklinikum Münster, Deutschland.