
Neue Herausforderungen in der Molekularpathologie von Lungentumoren
CME-Beitrag
Nach Jahrzehnten der entmutigenden Stagnation findet seit etwa 15 Jahren ein Paradigmenwechsel in der Behandlung früher prognostisch besonders schlechter solider Tumoren – wie des malignen Melanoms oder des nicht-kleinzelligen Lungenkarzinoms (NSCLC) – statt. Die Identifizierung genetischer Veränderungen in den zentralen Signalwegen der Tumorzelle auf genomischer und zunehmend auch epigenomischer Ebene hat die Form der Behandlung (i. v. Chemotherapie vs. orale Gabe von Tyrosinkinase-Inhibitoren) stark weiterentwickelt. Gleichzeitig haben sich Gesamtüberleben (OS), progressionsfreies Überleben (PFS) und die Lebensqualität deutlich verbessert. Viele Patienten, die unter diesen Tumoren leiden, können ambulant behandelt werden und haben statt einer Überlebenswahrscheinlichkeit von Wochen eine neue Perspektive von zum Teil mehreren Jahren.
Durch diesen Paradigmenwechsel hat sich in den letzten Jahren auch die Molekularpathologie grundlegend gewandelt. Je mehr Informationen dem Therapeuten vorliegen, desto gezielter kann der Krebs behandelt werden. Dazu sind solide und kosteneffektive Testmethoden nötig, die den hohen Anforderungen des diagnostischen Alltags genügen. Immer mehr Bioinformatik ist erforderlich, um technisch korrekte und wissenschaftlich gesicherte Befunde zu ermöglichen. Die Auswertung der Daten aus einem Multi-Gen-Panel ist deutlich aufwendiger als das bei herkömmlichen Methoden der Fall war. Jahrzehntelang bewährte diagnostische Nachweismethoden sind in relativ kurzer Zeit in manchen Bereichen obsolet geworden (z. B. die Sanger-Sequenzierung im Kontext der Liquid Biopsy-Testung). Unter dem Schlagwort Next Generation Sequencing (NGS) verbirgt sich mittlerweile ein ganzes Spektrum an verschiedenen Methoden mit unterschiedlicher Routinetauglichkeit. Dieser Beitrag nimmt zu aktuellen Entwicklungen im Bereich der Molekularpathologie Stellung.
Die Entwicklung der molekularen Lungendiagnostik in den letzten zehn Jahren
In den letzten Jahren hat die Beschreibung immer neuer Treibermutationen die Therapie und mit ihr die Diagnostik nicht nur des nicht-squamösen NSCLC revolutioniert. Während vor wenigen Jahren noch die Sanger-Sequenzierung als technische Basis in der Routinediagnostik akzeptiert war, gilt sie heute als weitgehend überholt. Dies hat mehrere Gründe. So ist die Nachweisgrenze von 20% Tumorallel bei der Sanger-Sequenzierung für einige Anwendungsgebiete (z. B.
Liquid Biopsy) nicht mehr ausreichend. Außerdem sind komplexere Fragestellungen hinzugekommen, beispielsweise bei Ovarialkarzinomen die BRCA1/2-Testung. Die beiden BRCA-Gene umfassen zusammen mehr als 5.000 Basenpaare und damit ca. 120 Amplikons (Sequenziereinheiten). Diese müssten mit der Sanger-Methode einzeln sequenziert werden, was für einen Patienten Wochen beanspruchen würde. Mittels NGS sind mehrere Gene dieser Größe problemlos und parallel bei mehreren Patienten mit relativ geringem Zeitaufwand sequenzierbar.
Speziell beim Lungenkarzinom ergeben sich weitere Herausforderungen für die molekulare Diagnostik. Häufig ist das Biopsiematerial in Größe und Tumorgehalt limitiert, d. h. es müssen möglichst sensitive Testmethoden zur Anwendung kommen. Außerdem sind die zu erwartenden genetischen Aberrationen relativ komplex, und enthalten neben Punktmutationen (z. B. im EGFR-, KRAS-, BRAF-Gen) auch Translokationen (von ALK-, ROS- oder RET-Gen) und Gen-Amplifikationen (von HER2, MET). Zum Nachweis der Punktmutationen wie auch kleinerer Insertionen und Deletionen kommen klassischerweise die Sanger-Sequenzierung oder auf Real-time-PCR basierende Plattformen wie z. B. der cobas®-Test zum Einsatz. Gen-Amplifikationen und Translokationen konnten bislang nur über FISH (Fluoreszenz-in-situ-Hybridisierung) bzw. Immunhistochemie (z. B. ALK) nachgewiesen werden. Eine relativ neue Form des NGS, das sogenannte Hybrid Capture NGS ermöglicht den zeitgleichen Nachweis aller genannten Aberrationen in einem Assay und in einem vertretbaren Zeitrahmen von etwa zehn Tagen. Eine vorgezogene Auswertung der wichtigsten Treiber-Gene und deren separate Befundung kann noch ein paar Tage in der aufwendigen Datenanalyse einsparen. Das Hybrid Capture NGS kann auf Formalin-fixiertes, in Paraffin eingebettetes (FFPE) Gewebe und auf zytologische Präparate angewendet werden und wird hier auch nach EBM-Ziffer vergütet; für die Liquid Biopsy ist dies bislang nicht der Fall.
Liquid Biopsy
Präanalytik
Unter dem Begriff Liquid Biopsy vereinen sich grundsätzlich zwei unterschiedliche Analyseformen. Zunächst ist es möglich, intakte, zirkulierende Tumorzellen (CTC, circulating tumor cells) aus dem peripheren Blut zu isolieren. Die zurzeit einzige von der FDA zugelassene Methode dafür ist das Cellsearch®-Circulating Tumor Cell Kit (Janssen Diagnostics Company, USA). Dieses Verfahren basiert auf dem Antikörper-vermittelten Nachweis von epithelialen Tumorzellen im Vollblut (CD45–/EpCAM+/Zytokeratin 8+/18+ und/oder 19+). Bei Krebspatienten konnte eine erhöhte Anzahl an CTC (> 5/ml) entitätsübergreifend mit einer ungünstigen Prognose korreliert werden [1–3]. Aufgrund der generell geringen Anzahl an CTC (0–10 CTC pro Milliliter Blut) ist eine stabile und routinetaugliche Mutationsanalyse allerdings schwierig und derzeit aufgrund der aufwendigen Methodik diagnostisch nicht relevant. Für das NSCLC ist Cellsearch® aufgrund eines zu geringen Detektionslimits auch gar nicht zugelassen. Trotz dieser Limitationen stellt diese Methodik sicherlich ein wichtiges Werkzeug in der grundlagenorientierten Tumorforschung dar.
Für diagnostische Zwecke weitaus zielführender ist die Mutationsanalyse an zellfreier zirkulierender Tumor-DNA (ctDNA, circulating tumor DNA). Diese wird von apoptotischen Tumorzellen in den Blutkreislauf abgegeben, hat eine Größe von etwa 170 Basenpaaren und kann prinzipiell zur Mutationsanalyse herangezogen werden. Dazu werden ein bis zwei Fixierungsröhrchen mit Vollblut benötigt (z. B. der Firma Streck oder Roche), die die Tumor-DNA stabilisieren. Das Prinzip stammt aus der Pränatal-Diagnostik und beruht auf drei Komponenten. Ein Fixierungs-Agens stabilisiert die Leukozyten und verhindert dadurch ein „Aussickern“ von Wildtyp-DNA. Tumor-DNA liegt im Blut unter Umständen in sehr niedriger Konzentration von weniger als 100 ng/µl vor, und eine weitere Verdünnung durch DNA aus normalen Körperzellen würde die Nachweisgrenze noch weiter herabsetzen. Weitere Reagenzkomponenten sind DNase-Inhibitoren und ein Anti-Koagulans.
Von klinischer Seite sind einige einfache, aber entscheidende Punkte bei der Blutabnahme zu beachten. Die Füllhöhe der Röhrchen (ca. 8 ml) sollte eingehalten werden; zu wenig Volumen führt zur Hämolyse, bedingt durch mechanische Beanspruchung beim Transport. Generell kann man die Röhrchen aber bei Raumtemperatur und bequem per Post verschicken; laut Hersteller ist die ctDNA in den Röhrchen für sieben Tage stabil. Das Blut darf nur mit einem dafür vorgesehenen Butterfly-Adapter (z. B. der Firma BD Biosciences) abgenommen und in die Vacutainer-Röhrchen gefüllt werden; dadurch soll ebenfalls eine Hämolyse verhindert werden.
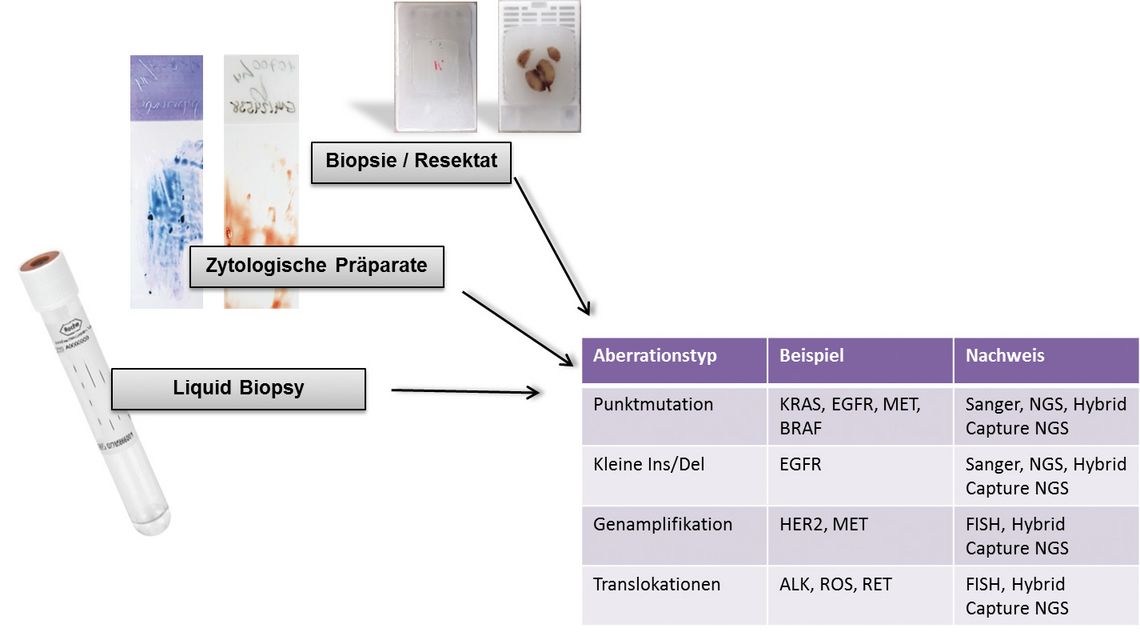
Indikationsstellung
Wir sehen seit 2015 eine starke Zunahme an Liquid Biopsy–Anforderungen. Den Großteil machen Rezidiv-Testungen aus; eine Primärdiagnostik findet bislang selten aus Blutplasma statt. Das Hauptproblem der Liquid-Biopsy-Testung ist und bleibt ihre eingeschränkte diagnostische Sensitivität. In aktuellen Studien liegt diese für das Lungenkarzinom bei etwa 70–80% [4]. Dies ist gleichbedeutend mit etwa 20–30% falsch-negativen Ergebnissen bei dieser Analyseform, weshalb die Gewebetestung weiterhin als Goldstandard anzusehen ist. Die Testung aus dem Plasma kann aber unter bestimmten Voraussetzungen sehr sinnvoll sein, nämlich dann, wenn der Patient unter bestehender Therapie mit Tyrosinkinase-Inhibitoren progredient ist und damit eine Therapieumstellung erforderlich wird. Wenn dann kein klassisches Tumormaterial gewonnen werden kann oder dies vom Patienten abgelehnt wird oder ihm nicht zugemutet werden kann, bietet die Plasmatestung eine minimal-invasive Alternative zur soliden Tumorbiopsie.
Ist keine Mutation im Blutplasma nachweisbar, so ist ein möglicher Grund dafür, dass keine ausreichenden Konzentrationen an Tumor-DNA in das Blut gelangen. Deshalb ist es in der Rezidivtestung auch wichtig, auf die ursprüngliche Mutation (z. B. EGFR-Exon-19-Deletion) zu testen. Ihr Vorhandensein kann beweisen, dass der Tumor generell in der Lage ist, ctDNA an das Blut abzugeben. Die Spezifität der Liquid-Biopsy-Testung liegt bei annähernd 100%. Dies bedeutet, dass eine EGFR-Mutation, die anhand der Liquid Biopsy bestimmt wird, mit hoher Wahrscheinlichkeit auch in der soliden Tumorbiopsie vorhanden ist.
Es gibt vermutlich eine Reihe biologischer Faktoren, die darüber bestimmen, ob ein Tumor ausreichende Mengen an ctDNA ans Blut abgibt (z. B. Größe, Vaskularisierung, Tumorlast, Lokalisation). Die damit einhergehenden Limitationen können auch durch noch so sensitive diagnostische Verfahren nicht ausgeräumt werden. Dennoch ist es wichtig, technisch möglichst ausgereifte, sensitive Testverfahren anzuwenden.
Die Sensitivität der Testung kann indirekt weiter durch klinische Parameter erhöht werden, indem Patienten mit entsprechend hoher Tumorlast oder schneller Progredienz getestet werden. Der Grund liegt darin, dass es im Einzelfall nicht vorhersehbar ist, wieviel ctDNA im Vergleich zu Wildtyp-DNA (aus apoptotischen gesunden Zellen oder Leukozyten) beim jeweiligen Patienten vorhanden ist.
Analytik
Deshalb ist die Sanger-Sequenzierung für den Mutationsnachweis an ctDNA ungeeignet; regionale und europäische Ringversuche hat diese Methode nicht bestanden. Daher sollten mindestens der cobas®-Test (Kit v 0.2) oder noch besser sensitive NGS-Plattformen (insbesondere Hybrid Capture) zum Einsatz kommen. Wir verwenden aktuell ein Hybrid-Capture-Verfahren mit einer Nachweisgrenze von 0,1% Tumorallel, womit eine Konkordanz zur herkömmlichen (nicht NGS-basierten) Gewebediagnostik von ca. 90% erreicht wird.
Eine weitere, noch empfindlichere Methode für den Mutationsnachweis im Plasma ist die digitale PCR (auch ddPCR, digital droplet PCR). Hier besteht allerdings wie bei allen PCR-Verfahren die Limitation darin, dass nur vorher definierte Mutationen detektierbar sind, weil jeweils spezifische Sonden eingesetzt werden müssen. Hinzu kommt eine relativ hohe Fehlerrate und ein technischer Aufwand, der den Einsatz zu Routinezwecken zurzeit (noch) begrenzt.
Weitere Indikationen
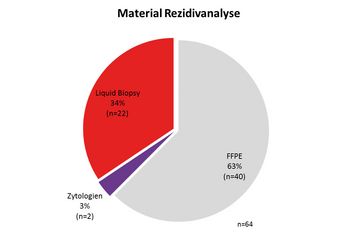
Wir sehen die Liquid Biopsy auch aufgrund immer höherer Genauigkeit in der Testmethodik weiter auf dem Vormarsch und haben bereits 2016 einen Anteil von 34% an der Rezidivtestung beim NSCLC beobachtet (Abb. 2).
In Zukunft ist der Einsatz der Liquid Biopsy durchaus auch zur Kontrolle der minimalen Resterkrankung (MRD) denkbar. Hierfür wäre eine regelmäßige (z. B. 3-monatliche) Plasmakontrolle auf Resistenzmutationen (z. B. EGFR T790M oder ALK L1196M) durchzuführen, um Informationen über eine etwaige genetisch begründbare Rezidivbildung zu erhalten, mit prädiktiven Aussagen zur Therapiewirksamkeit und bevor die klinische Symptomatik sehr ausgeprägt ist. Der dann voraussichtlich deutlich kleinere Tumor ließe sich vermutlich auch leichter kontrollieren.
Für die Pathologie ist allerdings noch ein neues Problem entstanden. Zwar ist das NGS nach langem Ringen seit Juli 2016 im neuen EBM-Katalog im Kapitel 19.4 („In-vitro-Diagnostik tumorgenetischer Veränderungen“) abgebildet. (Noch) nicht enthalten ist allerdings die Liquid Biopsy, unabhängig davon, mit welcher Methode getestet wird. Hier kann versucht werden, über eine Abtretungserklärung des Patienten und einen Kostenübernahmeantrag an die Krankenkasse eine Vergütung der Pathologie zu gewährleisten; immerhin belaufen sich zum Beispiel die Kosten einer EGFR-Testung an Plasma auf ca. 400 Euro.
Hot topic: mutational load
Die Einführung der NGS-Technologie hat zu ganz neuen diagnostischen Möglichkeiten geführt. Ein weiterer spannender Anwendungsbereich neben der Liquid-Biopsy-Testung ist die Bestimmung der Mutationslast des Tumors (Tumor Mutational Load, TML). NGS ermöglicht es dem Diagnostiker, die Anzahl der Mutationen in einer gegebenen Anzahl von Genen (und sogar im Gesamt-Exom) in einem realistischen Zeitrahmen zu bestimmen. Ein mögliches Anwendungsgebiet hierfür bietet die moderne Immuntherapie mit Checkpoint-Inhibitoren. Bei einem Anteil von ca. 20% der Patienten konnten damit langanhaltende Therapieerfolge beobachet werden. Seitdem wird intensiv versucht, diese Patientengruppe besser zu definieren, um prädiktive Marker für das Ansprechen auf Immunmodulatoren (z. B. therapeutische PD-1-/PD-L1-Antikörper) zu ermitteln.
Dabei war von Anfang an die Annahme naheliegend, dass Tumoren mit hoher PD-L1-Expression besonders gut auf PD-1-/PD-L1-Checkpoint-Inhibition ansprechen. Diese Erwartung hat sich zum Teil auch bestätigt; es gibt aber durchaus Gegenbeispiele, nämlich Tumoren, die trotz fehlender PD-L1-Expression gut auf diese Art der Immuntherapie reagieren. In der Zulassungsstudie für die Erstlinientherapie des metastasierten NSCLC mit dem Anti-PD-1-Antikörper Pembrolizumab konnte ein Überlebensvorteil für Patienten gezeigt werden, deren Tumorzellen zu mindestens 50% PD-L1 exprimieren [5]. Für den Anti-PD-1-Antikörper Nivolumab gibt es hingegen keine so klare Trennlinie; hier werden in unterschiedlichen Studien die Abstufungen ≥ 1%, ≥ 5% und ≥ 10% als Beurteilungskriterien herangezogen.
Zusammenfassend lässt sich sagen, dass die Immuntherapie mit Checkpoint-Inhibitoren im Prinzip funktioniert, aber nur für einen begrenzten Teil der Patienten und ohne die Möglichkeit, diese vorab eindeutig und ausreichend durch Biomarker zu definieren. Hier rückt die Tumor-Mutationslast (TML) als vielversprechendes zusätzliches Kriterium in den Fokus. Daten der Checkmate-026-
Studie zeigen, dass Lungentumoren mit hoher PD-L1-Expression (≥ 50%) und gleichzeitig hoher TML besonders gut auf PD-1-/PD-L1-Checkpoint-Inhibition ansprechen. Eventuell wird sich in Zukunft eine solche genetische Konstellation als besonders günstig für den Patienten herausstellen.
Besonders Erfolg versprechend scheinen Checkpoint-Inhibitoren bei kutanen Melanomen und bei mit Zigarettenkonsum assoziiertem NSCLC (v. a. Plattenepithelkarzinom) zu sein. Diese Entitäten zeichnen sich durch Hunderte bis Tausende von durch UV-Strahlung bzw. Tabak-Karzinogene induzierten Mutationen aus. Diese dürften zur Bildung einer Vielzahl von Neoantigenen führen und sollten daher besonders „fremdartig“ bzw. prinzipiell immunogen sein [6]. Tumoren mit geringerer Mutationslast sprechen dagegen schlechter auf eine Immuntherapie an (z. B. [7]).
Nun stellt sich die Frage, welche Gene für die Bestimmung der Mutationslast entscheidend sind und ab welcher Zahl von Mutationen man von einer signifikanten TML sprechen kann. Mehrere „Whole exome sequencing“(WES)-Studien haben kürzlich eine Korrelation zwischen dem „total mutational load“ und einem Therapieansprechen gezeigt [8, 9]. Das WES ist aber sehr aufwendig und teuer; Ziel muss es sein, eine geringere Anzahl von Genen zu ermitteln, aus deren Sequenzierung sich eine zielführende Aussage zur Mutationslast ableiten lässt. Zudem ist die Sequenzier-Tiefe beim WES für diagnostische Zwecke zu gering (ca. 30 vs. 5.000 reads). Es gibt Versuche, anhand kondensierter NGS-Panels praktikable Verfahren zu entwickeln (z. B. [10]). Es bleibt abzuwarten, ob sich in Zukunft eine Kombination aus dem TML und der PD-L1-Expression als sinnvoller prädiktiver Marker erweisen wird.
Neue Indikation: BRAF-Mutation beim NSCLC
Kürzlich wurde die Kombinationstherapie aus einem BRAF- und einem MEK-Inhibitor (Dabrafenib und Trametinib) für vorbehandelte NSCLC-Patienten zugelassen, die eine BRAFV600-Mutation aufweisen. Diese Kombination zeigte in einer Phase-II-Studie eine Gesamtansprechrate von 63%. Die Toxizität war vertretbar und das Therapieansprechen zum Teil länger andauernd [11]. Diese Kombination war zuvor schon für BRAFV600-mutierte Melanome zugelassen, nun profitiert also auch eine kleine Subgruppe der Patienten mit Lungenkrebs. Bei Lungenkarzinomen ist im Gegensatz zum Melanom nur die V600E-Mutation von BRAF beschrieben worden, nicht die V600K-Mutation.
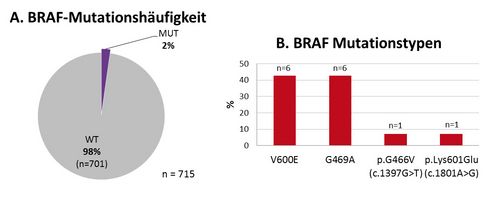
Seit längerer Zeit ist die BRAF-Testung in der Hämatopathologie Hamburg Bestandteil der Routinetestung. Früher wurde die Untersuchung mittels Realtime-PCR durchgeführt, heute wird dazu NGS verwendet. Die BRAF-Testung ist grundsätzlich auch an der Liquid Biopsy durchführbar. Die Gruppe von NSCLC-Patienten, die für die Kombinationstherapie mit BRAF- und MEK-Inhibitor infrage kommen, ist begrenzt. Unter den 2% der Patienten, die in unserem Kollektiv eine BRAF-Mutation aufwiesen (14/715), war nur bei sechs eine V600E-Mutation nachweisbar (Abb. 3). Internationale Studien bestätigen die relative Seltenheit der V600E.
Literatur
1. Cristofanilli M et al. Circulating tumor cells, disease progression, and survival in metastatic breast cancer. NEJM 2004; 351: 781-91.
2. Hu B et al. Circulating tumor cells in prostate cancer. Cancers 2013, 5(4), 1676-1690.
3. Muinelo-Romay L et al. Evaluation of Circulating Tumor Cells and Related Events as Prognostic Factors and Surrogate Biomarkers in Advanced NSCLC Patients Receiving First-Line Systemic Treatment. Cancers 2014, 6(1), 153-165.
4. Mok et al. Detection and Dynamic Changes of EGFR Mutations from Circulating Tumor DNA as a Predictor of Survival Outcomes in NSCLC Patients Treated with First-line Intercalated Erlotinib and Chemotherapy. Clin Cancer Res 2015; 21: 3196-203.
5. Reck M et al. Pembrolizumab versus Chemotherapy for PD-L1-Positive Non-Small-Cell Lung Cancer. N Engl J Med 2016; 375: 1823-33.
6. Alexander C et al. T-cell invigoration to tumour burden ratio associated with anti-PD-1 response. Nature 2016; 54: 60-5.
7. Overwijk WW et al. Mining the mutanome: developing highly personalized Immunotherapies based on mutational analysis of tumors. J Immunother Cancer 2013; 1: 11.
8. Champiat S et al. Exomics and immunogenics: Bridging mutational load and immune checkpoints efficacy. Oncoimmunology 2014; 3: e27817.
9. Roszik J et al. Novel algorithmic approach predicts tumor mutation load and correlates with immunotherapy clinical outcomes using a defined gene mutation set. BMC Med 2016; 14: 168.
10. Planchard D et al. Dabrafenib plus trametinib in patients with previously treated BRAFV600E-mutant metastatic non-small cell lung cancer: an open-label, multicentre phase 2 trial. Lancet Oncol 2016; 17: 984-93.


Dr. Markus Falk
Dr. Markus Tiemann
Hämatopathologie Hamburg
Fangdieckstr. 75A, 22547 Hamburg


