Multiples Myelom: Innovative Diagnostik in der klinischen Routine
Eine innovative molekulare Diagnostik beim Multiplen Myelom ermöglicht
1) eine Risikostratifikation und Erfassung therapeutischer Zielstrukturen, 2) die Einteilung in Subgruppen, 3) die Darstellung einer klonalen Heterogenität, und
4) eine Aussage über das Therapieansprechen (minimale Resterkrankung: vgl. gesondertes Kapitel). Daneben ist die molekulare Diagnostik entscheidend für das Verständnis der Pathogenese des Multiplen Myeloms sowie die Definition neuer Zielstrukturen.
Hintergrund ist, dass es sich beim Multiplen Myelom auf molekularer Ebene um eine vielschichtige Erkrankung handelt [1]. Wesentliche Methoden, die routinemäßig zum Einsatz kommen, sind die Interphase-Fluoreszenz-in-situ-Hybridisierung (iFISH; [2–4]) sowie an wenigen spezialisierten Zentren globale Genexpressionsanalysen (GEP) mittels Desoxyribonukleinsäure (DNA)-Microarrays [5–9]. Durch geeignete Analysemethoden (Genexpressionsreport, GEP-R) kann letztere auch in der klinischen Routine durchgeführt werden [10]. Experimentelle Methoden im Rahmen klinischer Studien sind die Array-komparative genomische Hybridisierung (aCGH), die mit einer Auflösung auf dem Niveau unterhalb einzelner Gene Veränderungen in der Kopienzahl (Zugewinne, Verluste) darstellen kann [11, 12], die RNA-Sequenzierung im Sinne einer Transkriptom-Analyse sowie die Exon- und Gesamtgenom-Sequenzierung. Letztere ermöglicht es, alle genetischen Veränderungen in Myelomzellen eines Patienten darzustellen [13–15].
Im Folgenden werden zunächst die Methoden beschrieben, die in die (erweiterte) klinische Routine Eingang gefunden haben, gefolgt von solchen, die noch als experimentell anzusehen sind. Im Anschluss hieran diskutieren wir deren klinischen, diagnostischen und therapeutischen Stellenwert. Wir schließen diesen Artikel mit einem Ausblick auf die zukünftige Entwicklung innovativer molekularer Diagnostik.
Methoden im klinischen Einsatz
Plasmazell-Aufreinigung
Hintergrund Das Knochenmark-Aspirat stellt auch bei einem hohen Plasmazell-Infiltrationsgrad ein Gemisch verschiedenster Zellpopulationen dar; die Gewinnung hochreiner Myelomzellen ist jedoch Voraussetzung für eine molekulare Charakterisierung des Myelomzell-Klons (Abb. 1).
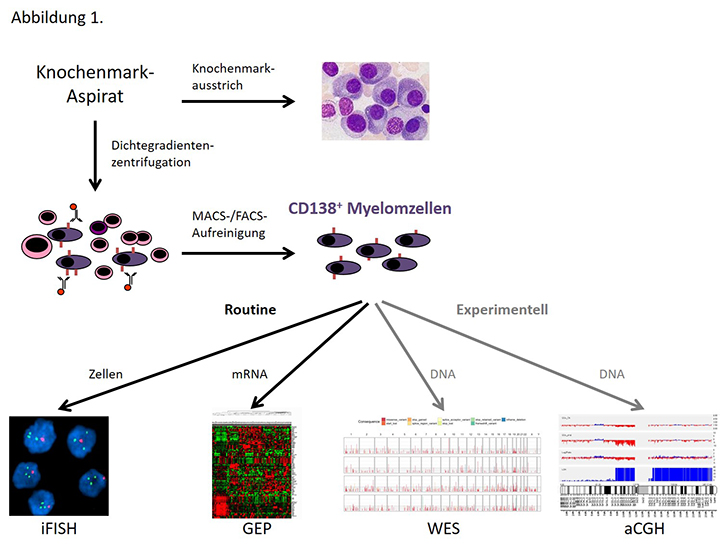
Methodik Aus 40–80 ml Knochenmark-Aspirat werden mittels Dichtegradienten-Zentrifugation mononukleäre Zellen isoliert. Anschließend erfolgt eine Positivselektion durch CD138-markierte Microbeads (magnetisch-aktivierte Zellsortierung) oder Fluoreszenz-markierte CD138-Antikörper (Fluoreszenz-aktivierte Sortierung). CD138 (Syndecan-1) ist ein für normale wie maligne Plasmazellen charakteristisches Oberflächenantigen.
Interphase-Fluoreszenz-in-situ-Hybridisierung (iFISH)
Hintergrund Die iFISH ermöglicht den Nachweis von numerischen Veränderungen einzelner chromosomaler Regionen (d. h. Veränderungen der Kopienzahl) sowie von strukturellen Veränderungen der Chromosomen (Translokationen). Die iFISH gehört zur Routinediagnostik bei Plasmazellerkrankungen.
Methodik Nach Zentrifugation CD138-aufgereinigter Plasmazellen auf einen Objektträger wird deren DNA denaturiert und mit fluoreszenzmarkierten, für eine bestimmte chromosomale Region spezifischen DNA-Sonden hybridisiert. Die Anzahl der gemessenen Hybridisierungssignale entspricht dabei der Anzahl an Kopien der jeweiligen Region (zwei Kopien: normal diploid, drei Kopien: Zugewinn, eine Kopie: Verlust). Die Fusion zweier Regionen, zum Beispiel von 11q13 und 14q32.3 bei einer Translokation t(11;14) (q13;q32.3), kann nachgewiesen werden, indem die beiden Regionen mit Sonden markiert werden, die mit unterschiedlichen Fluoreszenzfarbstoffen (z. B. rot und grün; Abb. 1) gekoppelt sind. Im Falle einer Fusion ergibt sich durch Überlagerung der Fluoreszenzen ein andersfarbiges, hier gelbes Fluoreszenzsignal.
Der Mindestsatz umfasst Sonden für t(4;14), Deletion 17p13 (del17p) und Zugewinn 1q21. Für die Untersuchung des Vorhandenseins subklonaler Aberrationen wird eine Myelomzell-Probe als eine subklonale Aberration enthaltend gewertet, wenn zumindest eine Aberration in
≥ 60% der Myelomzellen vorhanden ist und zumindest eine andere Aberration in 20–59% der untersuchten Myelomzellen detektiert wird.
Vorteil Relativ einfach durchzuführen, Erfassung der klonalen Heterogenität (Subklone).
Nachteil Nur eine begrenzte Anzahl an chromosomalen Regionen untersuchbar, interessierende Regionen müssen im Voraus definiert werden.
Preis 400–500 € je nach Sondensatz.
Methoden in der erweiterten klinischen Routine
Genexpressionsanalysen
Hintergrund Globale Genexpressionsanalysen mit kommerziellen Oligonukleotid-DNA-Microarrays ("Chips") wie beispielsweise Affymetrix U133 2.0 ermöglichen die Erfassung der Expression quasi aller menschlichen Gene (Transkriptom; vgl. Abb. 1).
Methodik Aus 20–100 ng der aus aufgereinigten Plasmazellen extrahierten Gesamt-RNA wird in zwei Amplifikationsschritten biotinylierte komplementäre Ribonukleinsäure (cRNA) hergestellt. Die markierte cRNA wird fragmentiert und mit Oligonukleotid-DNA-Microarrays hybridisiert. Im Anschluss daran werden die Microarrays gescannt und die relative Fluoreszenzintensität der einzelnen Gene (Probesets) gemessen. Die Fluoreszenz an diesem Ort ist dabei der Menge der gebundenen biotinylierten cRNA proportional, die ihrerseits wiederum mit der ursprünglich vorhandenen mRNA-Menge des betrachteten Gens korreliert.
Vorteil Erfassung der Expression
„aller“ Gene in einer Untersuchung (im Gegensatz zur PCR).
Nachteil Nur wenige Zentren haben hinreichend Erfahrung für den klinischen Einsatz.
Preis ca. 750 €.
Genexpressions-Report (GEP-R)
Hintergrund Wesentliches Element der risikoadaptierten bzw. personalisierten Therapie ist die Erfassung entsprechender Zielstrukturen und Realisierung der Risikostratifizierung innerhalb eines Zeitraumes, der es ermöglicht, klinische Konsequenzen umzusetzen (z. B. während des 1. Zyklus der Induktionstherapie).
Methodik Mit dem GEP-Report
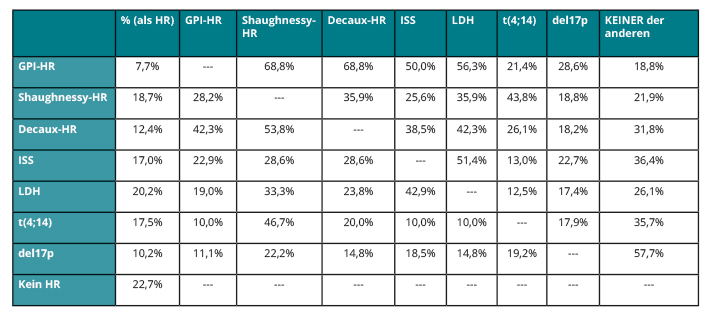
(GEP-R; [10]) haben wir eine nicht-kommerzielle, adaptierbare Software-Oberfläche entwickelt, welche die molekularen Klassifikationen [16, 17], Risikostratifikation [6, 18, 19] sowie die Erfassung von Zielgenen zur risikoadaptierten bzw. personalisierten Therapie in einen Befund integriert. Es wird eine Identitäts- sowie Qualitätskontrolle durchgeführt und der Befund anschließend als PDF-Dokument ausgegeben (Abb. 2). Um die Anwendung in der klinischen Praxis zu erleichtern, werden validierte klinische und Genexpressions-basierte Risikofaktoren in einem Metascore (HM-Metascore) zusammengefasst, der es dem Arzt erlaubt, eine prognostische Abschätzung abzugeben (Tab. 1).

Experimentelle Methoden
Array-komparative genomische Hybridisierung
Hintergrund Genomweite Veränderungen der Kopienzahl können mittels aCGH auf dem Niveau unterhalb einzelner Gene nachgewiesen werden. Methodenbedingt ist es nicht möglich, balancierte Translokationen (wie die meisten IgH-Translokation, z. B. t(4;14)) oder subklonale Aberrationen zu detektieren.
Methodik Verwendung finden in der Regel sogenannte Oligonukleotid-Arrays, zum Beispiel Affymetrix-SNP-(single nucleotide polymorphism)-Chips (Abb. 1).
Vorteil Nachweis genomweiter Veränderungen der Kopienzahl.
Nachteil Kein Nachweis balancierter Translokationen und subklonaler Veränderungen.
Preis ca. 500–1.000 €.
RNA-Sequenzierung
Hintergrund Die RNA-Sequenzierung ist eine neue Methode zum Erfassen des Transkriptoms, die sog. Next Generation Sequencing (NGS)-Technologien nutzt. Sie kommt 1. mit geringen RNA-Mengen aus (1 ng vs. 100 ng für „Chips“) und ermöglicht damit die expressionsanalytische Untersuchung früher Myelomstadien mit geringer Tumorlast. Sie ermöglicht 2. die Quantifizierung von Transkripten ohne signifikante Sättigungseffekte, setzt 3. keine A-priori-Definition von Sequenzen voraus und erlaubt die Erfassung von mutierten Transkripten und 4. die Analyse von Splice-Varianten sowie 5. im Prinzip die Untersuchung anderer RNA (z. B. micro-RNA).
Methodik Voraussetzung für die Anwendung von NGS-Methoden (s. u.) ist die Umschreibung der RNA in cDNA. Die bioinformatische Auswertung der Daten ist im Gegensatz zu GEP (z. B. mit GEP-R) aktuell noch sehr aufwendig und nicht standardisiert, weshalb die RNA-Sequenzierung im Moment überwiegend retrospektiv eingesetzt wird. Ziel unseres vom Bundesministerium für Bildung und Forschung geförderten Systemmedizin-Konsortiums CLIOMMICS ist es, basierend auf den Vorarbeiten zum GEP-R eine Translation der RNA-Sequenzierung in die klinische Routine zu ermöglichen.
Vorteil vgl. oben 1.–5.
Nachteil Keine Sättigung (daher tiefere Sequenzierung notwendig), komplexe Datenauswertung.
Preis ca. 500–1.000 €.
Sequenzierung: Gesamtgenom- und Exon-Sequenzierung
Hintergrund Die Gesamtgenom-Sequenzierung (whole genome sequencing, WGS) ermöglicht die Entschlüsselung der kompletten DNA-Sequenz, Exon-Sequenzierung (whole exon sequencing, WES) die Untersuchung der kodierenden Bereiche des Genoms und somit die Identifizierung erworbener somatischer Mutationen durch den Vergleich von Myelomzellen mit Normalgewebe des gleichen Patienten (Abb. 1).
Methodik NGS-Methoden, z. B. mittels Illumina HiSeq-Technologie, basieren auf einer Sequenzierung mittels DNA-Synthese, bei der pro Zyklus eine fluoreszenzmarkierte Base eingefügt und konsekutiv ausgelesen wird.
WGS
Vorteil Erfassung der kompletten DNA-Sequenz einschließlich Punktmutationen und struktureller Veränderungen.
Nachteil Sehr große Datenmengen, komplexe und langwierige Datenanalyse.
Preis ca. 5.000 €.
WES
Vorteil Zur Untersuchung der klonalen Heterogenität geeignet (tiefe Sequenzierung), im Vergleich zur WGS günstiger, kleinere Datenmenge.
Nachteil Lediglich Erfassung der kodierenden Bereiche des Genoms; kein Nachweis struktureller Aberrationen (z. B. Translokationen), große Datenmengen, aufwendige Datenanalyse.
Preis ca. 800 €.
Klinischer und therapeutischer Stellenwert
Molekulare Diagnostik – prognostischer und therapeutischer Stellenwert
iFISH Prognostisch relevant sind hier insbesondere das Vorliegen einer Translokation t(4;14), einer Deletion des kurzen Arms von Chromosom 17 (del17p) sowie von Zugewinnen des langen Armes von Chromosom 1 (1q-Zugewinn; [2, 4]). Ein alleiniger Verlust des langen Arms von Chromosom 13 (del13q), lange als wesentlicher prognostischer Faktor angesehen, gilt nicht mehr als solcher [4].
In Untersuchungen bei Patienten mit asymptomatischem, nicht therapiepflichtigem Myelom konnten wir zeigen, dass Patienten, bei denen eine der bereits beim therapiepflichtigen Myelom mit einer schlechten Prognose assoziierten chromosomalen Hochrisiko-Aberrationen (del17p, t(4;14) oder 1q-Zugewinn) vorliegt, eine signifikant schnellere Progression zur Therapiepflichtigkeit aufweisen als solche ohne die entsprechende Aberration [3]. Daneben konnten wir Hyperdiploidie als erstes Beispiel für einen adversen prognostischen Faktor beim asymptomatischen Myelom identifizieren, der beim symptomatischen Myelom mit einer neutralen Prognose einhergeht. Ein Teil der prognostischen Bedeutung chromosomaler Aberrationen ist somit nicht nur abhängig von der Therapie (entsprechend einem prädiktiven Faktor), sondern eine intrinsische Eigenschaft von Myelomzellen (und damit ein prognostischer Faktor im engeren Sinn). Dies ist insbesondere hinsichtlich von Versuchen relevant, negative prädiktive Faktoren durch eine spezifische Behandlung zu überwinden und kann umgekehrt deren Scheitern in molekularen Subgruppen erklären. Eine iFISH-Analyse ist bei geeigneter Logistik und Untersuchungstechnik bei über 95% der Patienten möglich und sollte für jeden Myelom-Patienten durchgeführt werden. Sie wird von den Krankenkassen übernommen.
GEP Mittels GEP können die Proliferationsrate der Myelomzellen [6], hochsignifikante Risikoscores (z. B. UAMS 70-Gen-Score, IFM 15-Gen-Score; [18 19] sowie einzelne prognostisch und ggf. therapeutisch relevante Gene erfasst werden [10]. Eine Genexpressionsanalyse ist bei geeigneter Logistik und Untersuchungstechnik auch im Rahmen von Multicenter-Studien bei ≥ 80% der Patienten möglich. Im Rahmen unseres vom Bundesministerium für Bildung und Forschung geförderten CAMPSIMM-Projektes konnten wir erfolgreich die klinische Einsetzbarkeit, d. h. Knochenmarkpunktion, Myelomzell-Aufreinigung, Durchführung der GEP und Erstellung des GEP-R innerhalb eines Zyklus Induktionschemotherapie, zeigen. Im Gegensatz zu Deutschland wird die GEP in den Vereinigten Staaten von einigen Krankenkassen übernommen.
Eine molekulare Charakterisierung mittels iFISH ist aktuell als Standard anzusehen. Zusammen mit der RNA- bzw. Exom-Sequenzierung wird sie in der Zukunft die Basis bilden für eine risikoadaptierte und ggf. personalisierte Therapie, insbesondere für Patienten mit ungünstiger Risikokonstellation oder solche im Rezidiv.
Schlussfolgerung für die Klinik
Die Möglichkeiten innovativer molekularer Diagnostik machen eine Zusammenarbeit von größeren universitären Zentren mit Kliniken und niedergelassenen Kollegen im Sinne einer optimalen Patientenversorgung mehr als wünschenswert.
Eine iFISH-Analyse sollte bei allen Patienten mit Multiplem Myelom durchgeführt werden.
Künftige Entwicklung
Prognostische Abschätzung
Methodisch betrachtet werden in den nächsten Jahren die iFISH sowie die GEP (mittelfristig ersetzt durch RNA-Sequenzierung) als klinische Werkzeuge eingesetzt werden. Beide Untersuchungen lassen sich in der Routine z. B. während des ersten Zyklus Induktionstherapie durchführen und somit zur Risikostratifizierung anwenden. Im Falle einer Hochrisikokonstellation könnte dann im Sinne einer risikoadaptierten Therapie die Therapie eskaliert werden. Wesentlich für ein solches Konzept ist jedoch ein signifikanter Hinweis, dass die entsprechende aggressivere Therapie auch die ungünstige Prognose eines oder einer Kombination von Faktoren aufhebt. Hier muss erneut betont werden, wie wichtig molekulare Untersuchungen sind, um entsprechende Informationen zu generieren.
Für die klinische Routine ist es zwingend, die Vielzahl prognostischer Faktoren in einen Score zusammenzufassen, um Arzt und Patienten eine verständliche Risikostratifizierung („sehr gut“, „gut“, „schlecht“, „sehr schlecht“) zu ermöglichen [10]. Hier wird es eine der wesentlichen Herausforderungen für die Zukunft sein, den prognostisch besten, aber gleichzeitig auch in der klinischen Routine bestimmbaren Metascore – in Abhängigkeit von den applizierten Wirkstoffen – zu entwickeln.
Neue Methoden werden bezüglich ihrer prognostischen Bedeutung in prospektiven klinischen Studien getestet; vor allem gilt dies gegenwärtig für die RNA- wie auch die Exon-Sequenzierung.
Therapeutische Konsequenzen
Personalisierte und risikoadaptierte Therapie
Mittels GEP bzw. Sequenzierung kann die Expression von Zielstrukturen nachgewiesen werden im Sinne einer personalisierten (nur Patienten, die die Zielstruktur exprimieren, werden mit dem entsprechenden Inhibitor behandelt) wie auch risikoadaptierten Behandlung (Patienten, die die entsprechende Zielstruktur exprimieren, haben eine ungünstigere Prognose). In ersten Untersuchungen bei Patienten mit Multiplem Myelom konnten mittels Sequenzierung Mutationen in Genen aufgedeckt werden, die bislang in der Myelom-Pathogenese noch unbekannt waren sowie Mutationen in Genen, gegen die es bereits Inhibitoren in der klinischen Testung gibt (z. B. BRAF-Inhibitoren; [20]). Die Erfassung aller chromosomalen Veränderungen einschließlich (exprimierter) Punktmutationen ermöglicht somit den Nachweis einer Vielzahl therapeutischer Zielstrukturen. Wesentlich ist hier, geeignete bioinformatische Auswertestrategien zu entwickeln, die einen Einsatz in der klinischen Routine ermöglichen.
Ein mögliches Studienkonzept stellt die personalisierte „Add-on“-Therapie dar. Denkbar ist, während des ersten Zyklus der Induktionstherapie eine Risikostratifizierung sowie die Erfassung von Zielstrukturen mittels iFISH und GEP bzw. Sequenzierung durchzuführen. In den folgenden Zyklen würde eine zusätzliche Gabe eines Inhibitors bei Expression der entsprechenden Zielstruktur evaluiert werden. Die Studie würde gegen die Induktionstherapie ohne Inhibitor-Zugabe randomisieren.
Aus pathophysiologischer Sicht ist die Frage interessant, ob unter verschiedenen Therapien eine lineare Entwicklung eines Myelomzell-Klones oder klonale Tiden unterschiedlicher Klone vorliegen. Erste experimentelle Daten zeigen eine klonale Heterogenität bei Diagnose wie auch das Auftreten unterschiedlicher klonaler Tiden zum Zeitpunkt der Krankheitsprogression nach erfolgreicher Therapie, letztere aufgrund des chemotherapeutischen Selektionsdruckes [21–23]. Angesichts des häufigen Vorhandenseins multipler (diskreter) fokaler Läsionen bzw. der diffusen Verteilung über das Knochenmark, die nicht im Knochenmark-Aspirat aus der Spina iliaca posterior superior erfasst sind, könnte zusätzlich zur temporalen eine räumliche klonale Heterogenität vorliegen [21, 24]. Auf Basis dieser Daten können wichtige klinische Konsequenzen gezogen werden [22]:
i) Sequenzielle Therapien mit Einzelsubstanzen sollten vermieden und stattdessen (möglichst) alle koexistierenden Subklone mit Kombinationstherapien eradiziert werden. Eine frühe suboptimale Behandlung könnte dagegen präferenziell einen eher indolenten Klon eradizieren und damit „Platz“ für einen aggressiveren Subklon schaffen. ii) Speziell in einem späteren Rezidiv kann, in Abhängigkeit von der Verfügbarkeit alternativer Therapielinien, eine Re-Exposition mit einer Therapielinie versucht werden, unter der ein Krankheitsprogress auftrat, wenn zwischenzeitlich unter einer anderen Therapie wieder ein sensitiver Subklon aufgetreten ist. iii) Ein teilweises Ansprechen (z. B. partielle Remission) muss nicht die partielle Suppression der gesamten Tumorzell-Population, sondern kann ebenso lediglich die eines sensitiven Klons bedeuten, während ein refraktärer Klon stabil bleibt und proportional dominanter wird.
Lokale Behandlung
Residuale Myelomzellen vor allem in osteolytischen bzw. fokalen Läsionen als Überlebensraum für Myelomzellen nach erfolgreicher Therapie i) bei Patienten, bei denen im sonstigen Knochenmark keine Myelomzellen mehr nachweisbar sind, bzw. ii) als Ursache der Progredienz von osteolytischen Läsionen und konsekutiv notwendiger (erneuter) chemotherapeutischer Behandlung sind putativ ursächlich dafür, dass fast alle Patienten einen Krankheitsprogress bzw. ein Rezidiv erleiden. Eine lokale Kontrolle dieser residuellen Myelomzellen könnte das Zeitintervall bis zu einer erneut notwendigen Chemotherapie verlängern bzw. zu einer Heilung des Myeloms in einer Subgruppe von Patienten beitragen. Eine Eradikation entsprechender Herde mittels Bestrahlung oder lokaler Therapie durch Wirkstoff-freisetzende Knochenersatzstoffe, wie sie z. B. im SFB Transregio TRR79 (http://www.uni-giessen.de/cms/ fbz/fb11/forschung/schwerpunkte/sfb/trr79) entwickelt werden (z. B. [25]), könnte eine Verlängerung des myelomfreien Überlebens und damit eine Heilung zumindest bei einem Teil der Patienten herbeiführen.
Literatur
1. Klein B et al. Molecular pathogenesis of multiple myeloma: Chromosomal aberrations, changes in gene expression, cytokine networks, and the bone marrow microenvironment. Recent Results Cancer Res 2011; 183: 39-86.
2. Avet-Loiseau H et al. Long-term analysis of the IFM 99 trials for myeloma: cytogenetic abnormalities [t(4;14), del(17p), 1q gains] play a major role in defining long-term survival. J Clin Oncol 2012; 30: 1949-52.
3. Neben K et al. Progression in smoldering myeloma is independently determined by the chromosomal abnormalities del(17p), t(4;14), gain 1q, hyperdiploidy, and tumor load. J Clin Oncol 2013; 31: 4325-32.
4. Neben K et al. Administration of bortezomib before and after autologous stem cell transplantation improves outcome in multiple myeloma patients with deletion 17p. Blood 2012; 119: 940-8.
5. Hose D et al. Induction of angiogenesis by normal and malignant plasma cells. Blood 2009; 114: 128-43.
6. Hose D et al. Proliferation is a central independent prognostic factor and target for personalized and risk adapted treatment in multiple myeloma. Haematologica 2011; 96: 87-95.
7. Hose D et al. Inhibition of aurora kinases for tailored risk-adapted treatment of multiple myeloma. Blood 2009; 113: 4331-40.
8. Seckinger A et al. Clinical and prognostic role of annexin A2 in multiple myeloma. Blood 2012; 120: 1087-94.
9. Seckinger A et al. Bone morphogenic protein 6: A member of a novel class of prognostic factors expressed by normal and malignant plasma cells inhibiting proliferation and angiogenesis. Oncogene 2009; 28: 3866-79.
10. Meißner T et al. Gene expression profiling in multiple myeloma – reporting of entities, risk and targets in clinical routine. Clin Cancer Res 2011; 17: 7240-7.
11. Avet-Loiseau H et al. Prognostic significance of copy-number alterations in multiple myeloma. J Clin Oncol 2009; 27: 4585-90.
12. Carrasco DR et al. High-resolution genomic profiles define distinct clinico-pathogenetic subgroups of multiple myeloma patients. Cancer Cell 2006; 9: 313-25.
13. Lohr JG et al. Widespread genetic heterogeneity in multiple myeloma: Implications for targeted therapy. Cancer Cell 2014; 25: 91-101.
14. Chapman MA et al. Initial genome sequencing and analysis of multiple myeloma. Nature 2011; 471: 467-72.
15. Walker BA et al. Mutational spectrum, copy number changes, and outcome: Results of a sequencing study of patients with newly diagnosed myeloma. J Clin Oncol 2015; 33: 3911-20.
16 Zhan F et al. The molecular classification of multiple myeloma. Blood 2006; 108: 2020-8.
17. Bergsagel PL, Kuehl WM. Molecular pathogenesis and a consequent classification of multiple myeloma. J Clin Oncol 2005; 23: 6333-8.
18. Shaughnessy JD et al. A validated gene expression model of high-risk multiple myeloma is defined by deregulated expression of genes mapping to chromosome 1. Blood 2007; 109: 2276-84.
19. Decaux O et al. Prediction of survival in multiple myeloma based on gene expression profiles reveals cell cycle and chromosomal instability signatures in high-risk patients and hyperdiploid signatures in low-risk patients: A study of the Intergroupe Francophone du Myélome. J Clin Oncol 2008; 26: 4798-805.
20. Andrulis M et al. Targeting the BRAF V600E mutation in multiple myeloma. Cancer Discovery 2013; 3: 862-9.
21. Egan JB et al. Whole genome sequencing of multiple myeloma from diagnosis to plasma cell leukemia reveals genomic initiating events, evolution and clonal tides. Blood 2012; 120: 1060-6.
22. Keats JJ et al. Clonal competition with alternating dominance in multiple myeloma. Blood 2012; 120: 1067-76.
23. Walker BA et al. Intraclonal heterogeneity and distinct molecular mechanisms characterize the development of t(4;14) and t(11;14) myeloma. Blood 2012; 120: 1077-86.
24. Zhou Y et al. Cytogenetic abnormalities in multiple myeloma: Poor prognosis linked to concomitant detection in random and focal lesion bone marrow samples and associated with high-risk gene expression profile. Br J Haematol 2009; 145: 637-41.
25. Striegler C et al. Dendritic glycopolymer as drug delivery system for proteasome inhibitor bortezomib in a calcium phosphate bone cement: First steps toward a local therapy of osteolytic bone lesions. Macromol Biosci 2015; 15: 1283-95.

(korrespondierende Autorin)
Dr. med. Anja Seckinger
E-Mail schreiben
Prof. Dr. sc. hum. Anna Jauch
Priv.-Doz. Dr. med. Dr. biol. hom. Dirk Hose
Universitätsklinikum Heidelberg,
Medizinische Klinik V
Im Neuenheimer Feld 410, 69120 Heidelberg
+49 6221 56 5427
+49 6221 56 6238

