NGS: Evidenz-basierte, medizinisch relevante Information
Diagnostik in der Onkologie
Automatisierte Arbeitsabläufe und komfortable Werkzeuge für die Auswertung genomischer Datensätze rücken das Next Generation Sequencing (NGS) zunehmend ins Blickfeld der Molekularpathologen. Auf der DGP-Jahrestagung 2016 stellte QIAGEN eine neue Gerätegeneration vor, die einen kompletten Workflow inklusive bioinformatischer Auswertung bietet.
Bei der 100. Jahrestagung der Deutschen Gesellschaft für Pathologie (DGP, Berlin) wurde deutlich, dass der analytische Trend in der onkologischen Molekularpathologie in Richtung Hochdurchsatz-Sequenzierung (NGS) geht. Diese innovative Technik ergänzt schon heute vielfach den Mutationsnachweis mittels Einzel-PCR, da sie zahlreiche diagnostisch, prognostisch und therapeutisch relevante Veränderungen in einem einzigen Arbeitsgang erfasst. Unterstützt wird die Einführung von NGS in die Routine durch kommerziell erhältliche Panels für relevante Mutationen. So ersetzt die Sequenzierung von EGFR, KRAS und ALK beim Bronchialkarzinom bis zu 20 herkömmliche PCR-Assays.
QIAGEN (Hilden, Deutschland) stellte auf einem Lunch-Symposium seine neue, mit einiger Spannung erwartete Lösung vor, die einen komplett integrierten Arbeitsablauf von der Probenvorbereitung bis zum Befund bietet (Abb. 1).
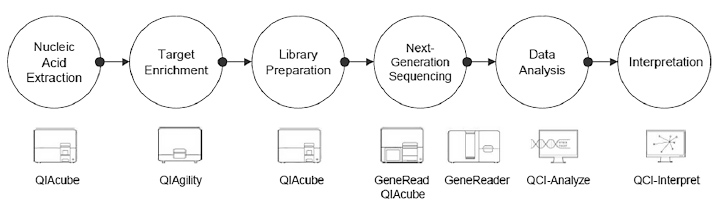
Das GeneReader NGS-System beherbergt vier voneinander unabhängige Sequenziereinheiten, die mit unterschiedlichen Serienlängen von 1 bis 40 Proben kontinuierlich beladen werden können.
Im Zentrum der Innovation steht aber nicht so sehr die Analysentechnik als vielmehr eine vollautomatische Auswertesoftware mit evidenz-basierter Interpretationsunterstützung unter Einsatz etablierter Datenbanken. Diese Bioinformatik-Plattform kombiniert die bislang größte Sammlung von (durch Experten kuratierten) wissenschaftlichen und klinischen Datenquellen, aktuellen Publikationen und klinischen Studien, internationalen Leitlinien und Empfehlungen sowie umfassenden Medikamenten-Informationen.
Das Endprodukt eines Sequenzierlaufs ist daher keine Liste von Genvarianten, sondern ein umfassender, klartextlicher Ergebnisbericht mit biochemischen und pathophysiologischen Erläuterungen aller gefundenen Mutationen, Angaben zu etablierten molekularen Therapieverfahren, vollständiger Bibliografie sowie – als besonderes Highlight – regelmäßig aktualisierten Hinweisen auf klinische Studien, in die die Patienten gegebenenfalls aufgenommen werden können.
Derzeit umfasst das vorgestellte GeneRead Actionable Insights Tumor-Panel
zwölf Gene; es erkennt in 330 Amplicons
773 Variantenpositionen mit nachgewiesener klinischer Bedeutung. Noch dieses Jahr soll die Palette durch Panels für Lungen-, Brust- und Ovarialkarzinome sowie eine umfassende BRCA-Mutationsanalytik (SNVs, INDELs und CNVs) ergänzt werden.
Ein weiteres kurzfristiges Ziel ist die Ausweitung des für NGS-Läufe einsetzbaren Probenmaterials in Richtung Liquid Biopsy. Erste Daten, die an Blutproben von NSCLC-Patienten erhoben wurden, lassen eine hohe Sensitivität von 1% erwarten. Auf der bevorstehenden ASCO-Konferenz (am.asco.org/) in Chicago soll erstmals der Workflow für die Flüssigbiopsie sowie ein Protokoll mit internen Kontrollproben für die qualitätsgesicherte Analyse von FFPE-Material vorgestellt werden.
Fazit
Auf dem Lunch-Symposium wurde offensichtlich kein weiteres Research-Gerät vorgestellt, sondern ein für die molekularpathologische Routine konzipiertes System, das bislang fragmentierte Arbeitsabläufe konsolidiert und Berührungsängste hinsichtlich der bioinformatischen Datenanalyse und Interpretation abzubauen hilft. Selbstverständlich bleibt auch hier die Befundverantwortlichkeit weiterhin beim Arzt, doch die evidenz-basierte Interpretationsunterstützung reduziert den benötigten technischen Wissensballast auf ein Minimum und ermöglicht eine Fokussierung auf die onkologisch relevanten Fragen.
Georg Hoffmann
Literatur
1. Oncology Insights Enabled by Knowledge Base-Guided Panel Design and the Seamless Workfl ow of the GeneReader NGS System. Qiagen White Paper 2016 (erhältlich auf Mail-Anfrage an Antje.Plaschke-Schluetter[at]qiagen[dot]com)
Lunch-Symposium "One Workflow, One Partner, Actionable Insights" im Rahmen der 100. Jahrestagung der DGP am 20. Mai 2016 in Berlin. Veranstalter: QIAGEN GmbH, Hilden.

