Möglichkeiten und Grenzen von Next Generation Sequencing (NGS) in der hämatologischen Diagnostik
Diagnostik in der Onkologie
Im Teil 1 dieser Darstellung zu aktuellen Strategien in der hämatologischen Diagnostik (Trillium Krebsmedizin 2016; 25: 120–23) wurden die seit Jahrzehnten bewährten Säulen der Zytomorphologie, der Histologie, der Immunphänotypisierung, der Zytogenetik, kombiniert mit Fluoreszenz-in-situ-Hybridisierung (FISH) und der Molekulargenetik dargestellt und miteinander in Bezug gesetzt. In Teil 2 soll es um die aktuellen Dimensionen und Perspektiven der Hochdurchsatz-Sequenzierungsverfahren, auch Next Generation Sequencing (NGS) oder Ultra-Deep Sequencing (UDS) genannt, gehen. Die technologische Entwicklung der letzten Jahre in diesem Bereich zeigt eine größere Geschwindigkeit als die in der Computertechnologie oder bei Kommunikationsplattformen wie Tablets oder Smartphones. Das heißt, dass uns mit den modernen Verfahren in der Forschung, aber eben auch in der Routinediagnostik (Zertifizierte Produkte, FDA-zugelassen, CE-zugelassen) ein zunehmend breiteres Portfolio von Möglichkeiten zur komplexen Sequenzierung von einzelnen Genen, Gen-Panels, ganzen Exomen (WES) oder Genomen (WGS) zur Verfügung stehen wird, das künftig prinzipiell auch im Rahmen des Einheitlichen Bewertungsmaßstabs (EBM) abrechenbar sein wird.
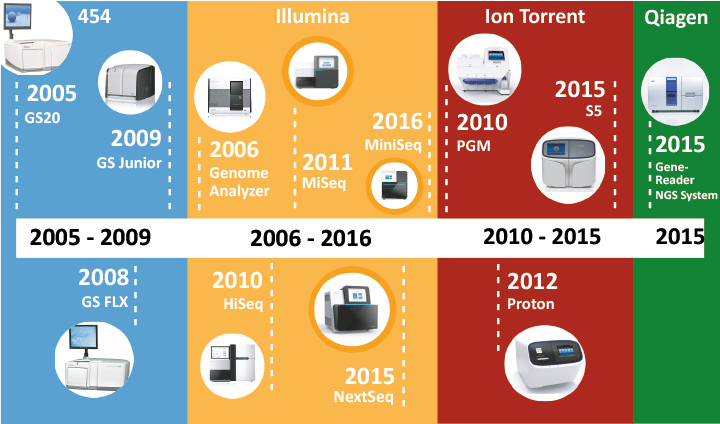
Die spezifischen Anforderungen an die hämatologische Diagnostik zum Auffinden individueller, spezifischer, genetischer und speziell molekulargenetischer Veränderungen bei der Diagnose, zur Prognose-Beurteilung und auch zum Nachweis einer minimalen Resterkrankung sind hoch. Moderne NGS-Geräte müssen sich deshalb bezüglich Sensitivität, Spezifität, Reproduzierbarkeit und Turn-Around-Zeit sowie Kosten diesen Ansprüchen stellen. Mehrere Anbieter haben in den letzten Jahren solide Plattformen zur Verfügung gestellt wie z. B. Roche/454, Ion Torrent/Thermo Fisher, Qiagen und insbesondere mit zunehmender Marktbeherrschung Illumina (Abb. 1).
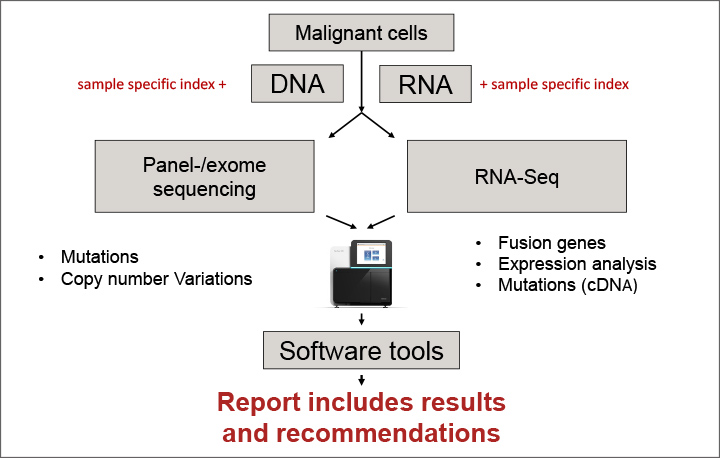
Die Fragen, die mit der NGS-Technologie gestellt werden können, sind vielfältig: Sie erfordern auf DNA ebenso wie auf RNA basierende Assays und sollen Fusionsgene und Copy-number-Veränderungen ebenso nachweisen wie Deletionen und Mutationen. Auch Genexpressions-Analysen bis hin zu DNA-Methylierungs-Assays sind möglich und gefordert. Zukünftige Workflows in der Molekulargenetik werden also zunächst durch die Hinzunahme von NGS komplex (Abb. 2).
Von zentraler Bedeutung ist die Aufgabenstellung, auf einer und derselben Maschine oder gar im gleichen Untersuchungslauf diese verschiedenen Fragestellungen zu bearbeiten und die parallele Prozessierung verschiedener Assays nicht nur technisch auf den Geräten, sondern auch durch verlässliche Software-Lösungen beim Read-Out abzubilden. Gerade der letzte Punkt, die Software-Lösungen und die Interpretation der erhobenen Daten, bleiben zurzeit deutlich hinter der rein technischen Entwicklung der Geräte zur Sequenzierung zurück. Schon die Verarbeitung der Datenmengen in akzeptabler Zeit (z. B. für die Routinediagnostik), insbesondere aber ihre Interpretation stellen eine immense Herausforderung dar und bergen zum jetzigen Zeitpunkt sicher auch Risiken. Vordringliche Aufgabe ist es also nicht nur, die NGS-Technologie für Forschung und Routinediagnostik weiter auszubauen und zur Anwendung zu bringen, sondern insbesondere auch die dabei erhobenen Daten mit sogenannten kuratierten Datenbanken (siehe unten) und einem laboreigenen Datenpool zum Abgleich zu bringen, um Fehlinterpretationen zu vermeiden.
Technische, ethische und finanzielle Dimensionen
Ziel soll hier deshalb nicht sein, noch einmal die verschiedenen Technologien in ihrer Unterschiedlichkeit zu beschreiben und zu bewerten, sondern vielmehr generell die Frage der Anwendbarkeit in der hämatologischen Routinediagnostik und deren technische, ethische und finanzielle Dimensionen zu erörtern.
In der Routinediagnostik müssen die genetischen Veränderungen zeitnah abgebildet werden: Nach wenigen Stunden bis einigen Tagen sollten die Ergebnisse in der Hämatologie, speziell bei akuten Leukämien, vorliegen. Voraussetzung ist eine Aufarbeitung der zu untersuchenden Zellen (z. B. peripheres Blut oder Knochenmark, auch Lymphknoten) nach Ficoll oder Lyse mit anschließender DNA- bzw. RNA-Präparation. In vielen Fällen ist vor der tatsächlichen Sequenzierung auf den NGS-Geräten eine Vorbereitung bzw. Anreicherung der informativen Gensequenzen mithilfe von Gen-Panels und einer sogenannten Amplikon-basierten Vorgehensweise notwendig.
Alternativ wäre an eine Whole-Exom- (WES) oder sogar eine Whole-Genome-Sequenzierung (WGS) zu denken. Diese beiden Szenarien können aus meiner Sicht in die aktuelle Routinediagnostik nicht eingeführt werden, v. a. aufgrund der immensen Datenpools, die dabei entstehen. Deren Verwendbarkeit und Interpretation sind so komplex, dass sie bei einer begrenzten Fragestellung in der hämatologischen Primärdiagnostik keine sinnvolle Basis darstellen. Für wissenschaftliche Fragestellungen ist das anders zu bewerten. Weiterhin ist der hohe Probendurchsatz nur bei wenigen Geräten mit entsprechend hoher Sequenzier-Kapazität möglich; das wird oft mit einer geringen Lesetiefe (Coverage) kompensiert und schließt die Detektion subklonaler Veränderungen praktisch aus. Der Nachweis geringer Tumorlasten, wie er in der Routinediagnostik erforderlich ist, verlangt aber gerade eine hohe Lesetiefe.
Eine Herausforderung:Aufbereitung und Interpretation der Daten
Nach Präparation der zu untersuchenden DNA- bzw. RNA-Bereiche, z. B. mithilfe von Gen-Panels in einer Größenordnung von je nach Fragestellung zwischen 20 und 100 Genen, erfolgt dann die Hochdurchsatz-Sequenzierung (Abb. 3).
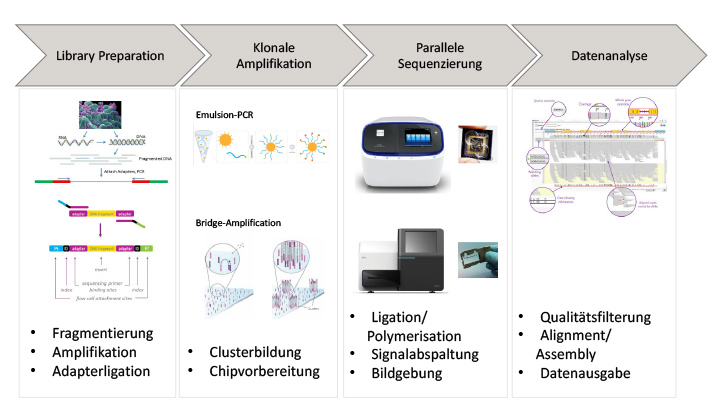
Die Laufzeit der Geräte liegt zwischen zehn und 40 Stunden, danach müssen die Daten aufbereitet, mit der Referenzsequenz abgeglichen und verrechnet werden. Anschließend erfolgt der Abgleich mit international verfügbaren Datenbanken zur Beurteilung der aufgefundenen jeweiligen „Varianten“ und die Bewertung des Ergebnisses durch z. B. Molekularbiologen, Humangenetiker, Hämatologen und/oder Pathologen. Eine nicht unerhebliche Herausforderung ist auch das Schreiben des Befundes, der internationalen Richtlinien entsprechen, aber ebenso für den einsendenden Arzt und für den betroffenen Patienten verständlich bleiben muss [1–3].
Das Wissen darüber, inwieweit eine im Genom gefundene Veränderung tatsächlich eine krankheitsauslösende Mutation darstellt, nimmt zu. Trotzdem gibt es neben zahlreichen Single Nucleotide Polymorphisms (SNPs) ohne pathogenetische Bedeutung auch sogenannte Varianten von unbekannter Signifikanz (VUS), deren tatsächliche Bedeutung für die Erkrankung oder den Patienten bisher nicht klar ist. Trotz Verwendung verschiedener Datenbanken und Software, die theoretisch auch die durch eine gefundene Mutation bedingte Veränderung der Proteinstruktur bewerten kann, bleiben hier bei dem zurzeit verfügbaren aktuellen Datenbestand Unsicherheiten. Dies gilt es zunächst zu realisieren und natürlich in der täglichen Arbeit der Routinediagnostik zu berücksichtigen.
Ungeklärte ethische Fragen
Ein weiterer Punkt sind die vielen ungeklärten ethischen Fragen zur Verwendung bzw. Nichtverwendung der Daten. Wie soll mit Daten verfahren werden, die im Rahmen eines angewendeten Assays erhoben wurden, primär aber gar nicht gefragt waren? Sollen diese Daten insgesamt gar nicht erst ausgewertet werden, sollen sie vom Gerät gar nicht erst ausgegeben werden oder ist es ethisch und ärztlich notwendig, auf alle Daten einer Analyse zu schauen, auch wenn nur ein Teil primär für sinnvoll erachtet wurde? Wie verfährt man mit Veränderungen in Genen, die primär nicht im Fokus der jeweiligen Analyse standen, aber von entscheidender diagnostischer oder therapeutischer Konsequenz sein können? Schon bei einer Panel-Sequenzierung mit einer überschaubaren zweistelligen Zahl von Genen stellen sich diese Fragen, für die Anwendung einer Exom- oder Genom-Sequenzierung in der hämatologischen Routinediagnostik ist dies momentan noch überhaupt nicht zu beantworten [4, 5].
Erfreulicherweise sind zu diesen ethischen Punkten der Verwendbarkeit von NGS-Daten an verschiedenen Stellen Diskussionen angeregt worden, die auch schon zu Thesenpapieren geführt haben [2, 3]. Dies in die Diagnostik umzusetzen, wird aber sicher noch sehr viel Zeit und auch Schulung der im Labor arbeitenden Leistungserbringer ebenso wie der Ärzte und letztlich auch der Patienten erfordern.
Außerdem ist darauf hinzuweisen, dass gerade auch die NGS-Technologie strenge Qualitätsanforderungen im Labor erfüllen sollte: Eine Akkreditierung ist für die Routinediagnostik möglich und notwendig. In den letzten Jahren sind verschiedene internationale Richtlinien zur Verwendung von NGS sowohl in technischer Hinsicht als auch die Auswertung der Daten bis hin zum Befund betreffend erschienen. Es gibt auch eine Diskussion, die durch die US-amerikanische Food and Drug Administration (FDA) sowie durch Arbeitsgruppen im Bereich der EU gesteuert wird. Es ist jedem, der in die NGS-basierte Diagnostik in der Hämatologie einsteigen möchte, dringend zu empfehlen, sich hier vorher tiefgreifend zu informieren und die schon zur Verfügung stehenden Richtlinien und Empfehlungen zu beachten.
Anwendbarkeit von NGS-Methoden neu geregelt
Seit Oktober 2013 war die Verwendung der NGS-Technologie zur Erbringung von abrechnungsrelevanten Leistungen analog bzw. innerhalb des EBM explizit untersagt. Diese Richtlinien wurden jetzt inhaltlich und technisch und mit Festlegung von Abrechnungsmodalitäten überarbeitet und werden einschließlich der Angaben zur Anwendbarkeit der NGS-Technologie zum 1. Juli 2016 in Kraft treten (Deutsches Ärzteblatt, Jahrgang 113, Heft 16, 22.04.2016).
Da sich die NGS-Technologie als Methode der molekularen Diagnostik im Umfeld von Zytogenetik und FISH darstellt, erfolgte hier eine umfassende Überarbeitung aller zytogenetischen und molekulargenetischen Testverfahren mit teilweiser Neubewertung. In diesem Bereich kann die NGS-Technologie jetzt auch eingesetzt werden. Dabei sind die verschiedenen Möglichkeiten (Nachweis von Mutationen, Sequenzierung einzelner Gene, Sequenzierung von Gen-Panels) geregelt. Es wird als Voraussetzung auch die „Prüfung der Indikationsstellung“ durch dazu berechtigte Ärzte festgeschrieben. Das Gleiche gilt für das Schreiben der zunehmend komplexeren Befunde. Damit wird zurecht ein hoher Standard an die Erbringung dieser Leistungen angelegt.
In der hämatologischen Diagnostik sind im EBM weiterhin die Verwendung von Whole-Genome- oder Whole-Exome-Sequenzierungen in der Routine ausgeschlossen. Eine Notwendigkeit dafür mag in Einzelfällen bestehen, in denen die Durchführung dann vorab zu beantragen ist.
Anforderungen an die Sensitivität erzwingen Umstieg auf NGS-Methodik
Die Routineuntersuchungen werden gestaffelt nach der Länge der zu sequenzierenden Gene bzw. Genabschnitte festgelegt und nach oben gedeckelt bzw. abgestaffelt. Diese Regelung ist diskussionswürdig, sie scheint beim aktuellen Stand der technischen Entwicklung und der Diskussion zwischen den Krankenkassen und den kassenärztlichen Vereinigungen aber die einzige kompromissfähige Lösung zu sein. Übergreifend für alle Sequenzierungstechniken wird darauf hingewiesen, dass ab April 2017 mit den jeweils verwendeten Methoden/Assays eine Sensitivität ≤ 10% erreicht werden muss. Dies ist nach allgemeiner Ansicht von Experten mit der zurzeit aktuellen, sogenannten Sanger-Sequenzierung nicht erreichbar, deren Sensitivität zwischen 10% und 20% anzusiedeln ist. Die beschlossenen Änderungen im EBM setzen also voraus, dass mittel- bis kurzfristig insgesamt auf NGS-Technologien umgeschwenkt werden muss, die zum jetzigen Zeitpunkt in der Regel eine Sensitivität zwischen 1% und 2% erreicht.
Des Weiteren ist im Bereich der Molekulargenetik auch der Nachweis der minimalen Resterkrankung zur Therapiesteuerung geregelt. Auch wird darauf hingewiesen, dass für den spezifischen Einsatz einzelner zielgerichteter Medikamente (personalisierte Medizin) die dabei im Vorfeld zu testenden genetischen bzw. molekulargenetischen Veränderungen gezielt getestet und auch in speziell dafür eingerichteten Unterkapiteln abgerechnet werden können und sollen.
Es bleibt abzuwarten, inwieweit sich die einzelnen Leistungserbringer (Humangenetiker, Pathologen, Hämatologen, Laborärzte) mit den hier neu definierten Testverfahren und Abrechnungsmöglichkeiten an einer zukunftsorientierten Diagnostik, die NGS-basiert sein wird, beteiligen können. Auch wenn möglicherweise mittelfristig die Anschaffungskosten für Geräte und die Kosten pro Analyse weiter fallen werden, bleiben nach wie vor ungeklärt die Kosten für die bioinformatische Datenanalyse und den Datenabgleich mit internationalen Datenbanken sowie für die Erstellung eines Befundes unter Abwägung der Datenqualität, der molekularbiologischen Implikationen und der sich daraus ableitenden ärztlichen, therapiesteuernden Konsequenzen. Hier ergeben sich komplett neue Felder der ärztlich-ethischen Verantwortung für unsere Patienten, deren Abbildung im EBM allerdings zeitnah geklärt werden muss, um die vorhandenen Möglichkeiten der modernen Diagnostik und die zunehmenden Möglichkeiten einer modernen, zielgerichteten Therapie auch umsetzen zu können.
Zusammenfassung
Zusammenfassend steht die Einführung von NGS in die Routinediagnostik in der deutschen Hämatologie technisch und auch abrechnungstechnisch kurz bevor. Auch wenn die aktuelle Gerätegeneration der verschiedenen Anbieter hier sehr verlässliche Daten liefern kann, ist die zur Dateninterpretation und zur Befunderstellung vorhandene Software-Landschaft bei Weitem noch nicht ausgereift genug, um das NGS zu einem diagnostischen Werkzeug für „jedermann“ zu machen. Neben der großen Datenmenge und der Komplexität ihrer Interpretation stellen sich auch bisher unbeantwortete ethische Fragen zur Verwendung der Daten und deren Speicherung. Die Beschäftigung mit diesen offenen Punkten wird sicher noch einige Jahre dauern, stellt aber eine lösbare Aufgabe dar.
Dessen ungeachtet werden sich die neuen Entwicklungen aber zeitnah nicht nur in eine bessere Diagnostik, sondern auch in gezieltere Therapieempfehlungen umsetzen lassen. Die Bündelung verschiedener Assays und Fragestellungen auf den neuen Plattformen der NGS-Technologie, eine möglichst weitgehende Automatisierung und eine Akkreditierung der NGS-Technologie wird die Diagnostik in der Hämatologie schneller, valider und Patienten-spezifischer machen.
Es muss das vorrangige Ziel aller Ärzte und im Labor in der Diagnostik beschäftigten Kolleginnen und Kollegen sein, hier eine offene und zielorientierte, am Patienten ausgerichtete Diskussion zu führen, um rasch die technischen Möglichkeiten auch in therapeutische Fortschritte umzusetzen. Ordnende und Perspektiven aufzeigende Vorreiterrollen nehmen hier z. B. der Arbeitskreis Laboratorium in der DGHO, die American Society of Hematology (ASH) mit ihrer neu eingerichteten „Task Force for Precision Medicine“ und auch EU-eigene Institutionen für personalisierte Medizin ein. Die Krankenkassen werden das ihre tun müssen, um nicht hinter den technischen Möglichkeiten, die jedem einzelnen Patienten zugutekommen können, zurückzubleiben: Die Kosten für Diagnostik machen im Vergleich zu Medikamentenkosten einen sehr kleinen Anteil der Kosten im Gesundheitswesen aus. Eine gezielte hämatologische und onkologische Therapie unserer Patienten benötigt zuvor eine molekularbasierte Routinediagnostik.
Literatur
1. Rehm HL et al. ACMG clinical laboratory standards for next-generation sequencing. Genet Med 2013; 15: 733-47.
2. Matthijs G et al Guidelines for diagnostic next-generation sequencing. Eur J Hum Genet 2016; 24: 2-5.
3. Aziz N et al. College of American Pathologists' laboratory standards for next-generation sequencing clinical tests. Arch Pathol Lab Med 2015; 139: 481-93.
4. Projektgruppe EURAT. Eckpunkte für eine Heidelberger Praxis der Ganzgenomsequenzierung. Heidelberg, 2015.
5. Schwarzkopf G. Ethische Grundfragen der Genomsequenzierung. Nova Acta Leopoldina 2013; 396: 9-24.
Prof. Dr. med. Dr. phil. Torsten Haferlach
MLL, Münchner Leukämielabor GmbH
Max-Lebsche-Platz 31, 81377 München
+49 89 99017100
+49 89 99017109
torsten.haferlach[at]mll[dot]com
www.mll.com

