Hämatologische Stufendiagnostik: Auf geradem Weg von der Morphologie zum Next Generation Sequencing?
Torsten Haferlach
Die hämatologische Diagnostik verwendet mehrere grundlegende Methoden, die sich schrittweise ergänzend auseinander ergeben können, aber auch als Einzelmethoden entscheidende Informationen liefern. Es ist zwischen methodischem Vorgehen bei der Primärdiagnose und der Verlaufsuntersuchung zu unterscheiden. Das Portfolio der zur Anwendung kommenden Analysemöglichkeiten besteht aus: Zytomorphologie, ergänzt durch Zytochemie, Histologie ergänzt durch Immunhistologie, klassische Chromosomenanalyse, Fluoreszenz-in-situ-Hybridisierung, Immunphänotypisierung (synonym FACS [Fluorescence-activated cell sorting] oder FLOW [Flow cytometry = Durchflusszytometrie]) und einer Vielzahl von molekularen Assays (Abb. 1; [1]). Bei letzteren hat sich die Methode des sogenannten Next Generation Sequencing (NGS), auch Hochdurchsatz-Sequenzierung genannt, in den letzten Jahren schrittweise von einem explorativen Werkzeug zu einer routinetauglichen Methode entwickelt, deren Stellenwert zu definieren ist [2]. Für die Bestimmung der minimalen Resterkrankung (MRD) bei Verlaufsanalysen sind speziell die Immunphänotypisierung und die molekulargenetischen Methoden von Bedeutung [3]. Für eine sinnvolle Stufendiagnostik gilt es jetzt, für die einzelnen Methoden den richtigen Stellenwert und die richtige Reihenfolge zusammen mit der klinischen Anwendbarkeit abzuwägen und zukunftsorientiert zu definieren.
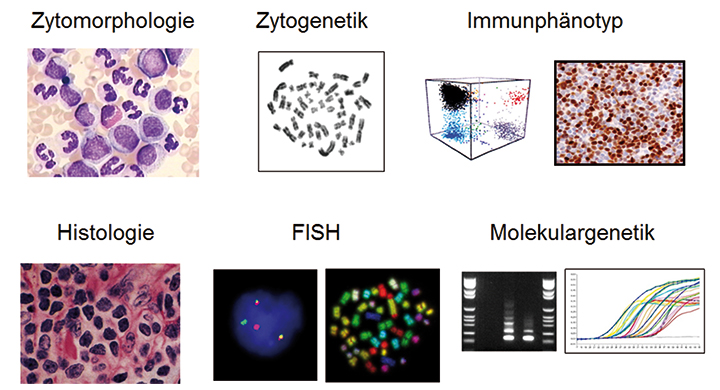
Zytomorphologie, Histologie
Die Untersuchung von Blut- und Knochenmark-Material ist seit mehr als einem Jahrhundert die Basis der hämatologischen Stufendiagnostik und wird dieses bis auf Weiteres auch bleiben. Die kenntnisreiche Analyse des Differenzialblutbildes (zum Teil unterstützt von zunehmend besseren Laborgeräten, zum Teil auch digitaler Bilderkennung) und – bei Auffälligkeiten – von Knochenmarks-Proben sind der Goldstandard. In Ergänzung zur klassischen Pappenheim-Färbung werden die Myeloperoxidase, die unspezifische Esterase und auch die Eisen-Färbung als obligat angesehen. Letztere ist insbesondere in der Diagnostik der myelodysplastischen Syndrome (MDS) für Diagnose und Klassifikation, zum Teil auch für die Therapieentscheidung notwendig. Bei auffälligen Blutbildwerten gilt es, eine Knochenmark-Punktion (Aspiration) und -Histologie (sogenannte Beckenstanze) anzuschließen. Das gewonnene Material wird mit verschiedenen Färbereaktionen und bei der Beckenstanze zum Teil immunhistologisch untersucht. Die dabei notwendigen Differenzierungen von peripheren Blut- und Knochenmarkzellen sowie zum Beispiel auch die Beurteilung der Fibrose und der Knochenmark-Architektur in der Histologie sind von grundlegender Bedeutung in der hämatologischen Diagnostik und steuern in vielen Fällen alle anderen Methoden. Da es sich dabei vielfach um „phänotypische Besonderheiten“ handelt, setzen diese Verfahren eine große Erfahrung und Expertise mit Kenntnissen zur Klinik des Patienten (Anamnese!) und den potenziell sich anzuschließenden weiteren Methoden voraus. Dieses Wissen ist die Basis einer soliden, kosteneffektiven und patientenorientierten Diagnostik in der Hämatologie [4].
Klassische Chromosomen-Analyse, Zytogenetik
In der hämatologischen Diagnostik spielen Chromosomen-Veränderungen sowohl zur Diagnostik und Klassifikation als auch zur Prognoseabschätzung eine zentrale Rolle. Bei Nachweis von malignen Zellen im peripheren Blut kann eine Chromosomen-Analyse auch aus peripherem Blut versucht werden. Ansonsten ist Material aus der Knochenmark-Aspiration (mit Heparin antikoaguliert) für die Zellkulturen notwendig. Die Ergebnisse werden nach internationalen Regeln der Zellkultivierung und insbesondere auch der Nomenklatur für die festgestellten Aberrationen (ISCN) erstellt. Diese Methode, die auf vitale Zellen angewiesen ist, kann nur bei guter Materialentnahme, kurzen Versandzeiten zum Labor (bis zu 48 Stunden bei Raumtemperatur) und von in der Tumor-Zytogenetik erfahrenen Laboren die bestmöglichen Ergebnisse liefern [5].
Fluoreszenz-in-situ-Hybridisierung (FISH)
Mithilfe von genspezifischen oder chromosomenspezifischen Fluoreszenz-Sonden kann man sowohl an Interphase-Zellkernen als auch an Metaphasen (aus der oben beschriebenen klassischen Chromosomen-Analyse, Zytogenetik) bestimmte Zielstrukturen detektieren. Im Gegensatz zur klassischen Chromosomen-Analyse, wo das gesamte Chromosomen-Material beurteilt werden kann, ist hier die Fragestellung vorher zu definieren, damit die entsprechenden Gensonden ausgesucht werden können. Diese Methode wird in Ergänzung zur klassischen Chromosomen-Analyse zur Aufklärung komplexer Strukturen verwendet, ebenso wie zur Bestätigung von chromosomalen Veränderungen der klassischen Zytogenetik. Die dabei definierten veränderten Gene oder Chromosomen (zum Beispiel Zugewinn oder Verluste) ergänzen die Diagnostik ebenso wie die Klassifikation und sind auch als Technik für die Verlaufsanalysen, d. h. zur Bestimmung der minimalen Resterkrankung verwendbar. Im Gegensatz zur oben genannten klassischen Chromosomen-Analyse sind die mittels FISH möglichen Untersuchungen an den Interphase-Zellkernen nicht auf vitale Zellen angewiesen und somit auch unabhängig von klassischer Zytogenetik anwendbar, solange die Fragen beziehungsweise die zu untersuchenden Zielstrukturen bekannt sind. Ein Gesamtscreening auf zytogenetische Veränderungen mit dieser Methode ist allerdings nicht sinnvoll.
Multiparameter-Immunphänotypisierung
Die heutigen technischen Möglichkeiten mit fünf bis zehn Farben zur Immunphänotypisierung lassen komplexe Analysen im Bereich der Leukämie- und Lymphomdiagnostik in kurzer Zeit (innerhalb eines Tages) zu. Die verwendeten Antikörper-Panels (CD) sind entsprechend der Verdachtsdiagnose zusammenzustellen. Deswegen sind gerade auch hier die Vorkenntnis der Klinik und die klinische Fragestellung sowie die Befunde aus peripherem Blut und Knochenmark im Rahmen einer Stufendiagnostik von größter Bedeutung. Im Bereich der akuten Leukämien, aber auch gerade bei den Lymphomen spielt die Immunphänotypisierung, zusammen mit der Immunhistologie, eine entscheidende Rolle zur Subklassifikation der jeweiligen Entitäten und hat große diagnostische und therapeutische Relevanz. Das dazu zu untersuchende periphere Blut oder Knochenmark sollte innerhalb von 48 Stunden analysiert werden, um aussagekräftige Ergebnisse zu ermöglichen. Die Methode ist für die Primärdiagnose ebenso wie für die Bestimmung der minimalen Resterkrankung, zum Beispiel durch die Definition des Leukämie-assoziierten Immunphänotyps (LAIP), geeignet [6, 7].
Molekulargenetik
In den letzten zehn Jahren haben sich die Möglichkeiten und Notwendigkeiten der molekularen Analysen in der hämatologischen Diagnostik bahnbrechend erweitert. Das gilt sowohl für die methodischen Aspekte als auch für die damit erstmals beschriebenen mutierten Gene. Durch die Verwendung zum Beispiel von Whole Genome Sequencing (WGS) oder Whole Exome Sequencing (WES) sowie durch Genexpressionsanalysen ist der Wissenszuwachs in der Molekulargenetik gerade in der Hämatologie immens [8]. Dieses muss in der hämatologischen Diagnostik im Rahmen einer Stufendiagnostik abgebildet werden, um die Analysen möglichst zielgerichtet, kostengünstig und erfolgreich einsetzen zu können.
Dabei stellen die mit molekulargenetischen Methoden zu untersuchenden Zielstrukturen in der Primärdiagnostik wichtiges Wissen über die Erkrankung zur Verfügung, haben zunehmend therapeutische Konsequenzen im Hinblick auf zielgerichtete Therapieansätze und sind auch als Marker im Verlauf zur Therapiesteuerung von immer größer werdender Bedeutung [9, 10].
Die dabei verwendeten molekularen Methoden wie zum Beispiel die klassische sog. Sanger-Sequenzierung, Schmelzkurven-Analytik, Fragmentanalyse, Real-Time-PCR (Polymerase-Kettenreaktion), Genexpressionsanalysen und in letzter Zeit verschiedene Methoden des sogenannten Next Generation Sequencing (NGS) stellen hohe Anforderungen an die untersuchenden Labore. Dies gilt nicht nur für die Qualitätssicherung im Bereich der verschiedenen Analysemethoden und der zu untersuchenden Zielstrukturen (Ringversuche, Akkreditierung), sondern auch für die Datenqualität und -interpretation. Die Vielzahl der jetzt bekannten und noch weiter entdeckten molekulargenetischen Veränderungen, die auf ihre tatsächliche pathophysiologische Bedeutung zu prüfen sind, setzt zunehmend ein erfahrenes Team von Molekularbiologen, Hämatologen und Pathologen und auch biomathematischer Expertise voraus. Gerade bei Verwendung von Next Generation Sequencing (Abb. 2) mit großen, bisher ungeahnten Datenmengen und -informationen müssen diese Ergebnisse mit großer Sorgfalt ausgewertet, interpretiert, mit internationalen Datenbanken abgeglichen und letztlich als Befund für den behandelnden Arzt und den Patienten zum „Sprechen“ gebracht werden [11].
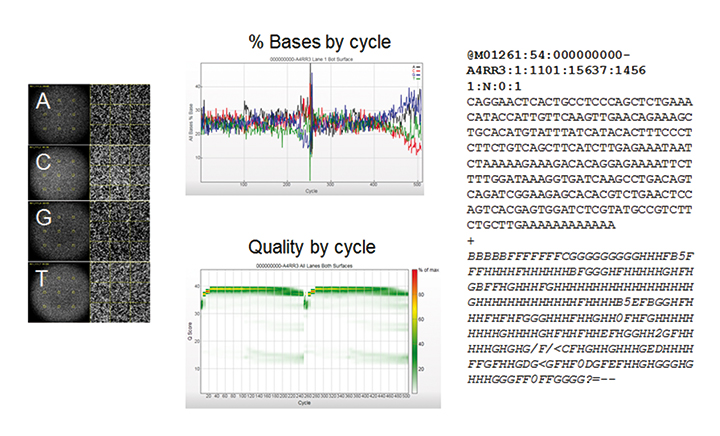
Dies macht es umso wichtiger, molekulargenetische Untersuchungen im Sinne einer Stufendiagnostik, ausgehend von den phänotypischen Befunden aus peripherem Blut und Knochenmark und der klinischen Anamnese – gegebenenfalls auch erst nach Vorliegen von zytogenetischen Befunden und den Ergebnissen der Immunphänotypisierung beziehungsweise Immunhistologie – einzusetzen. Möglich wäre ein solches Vorgehen bei unklaren Befunden, da für molekulare Analysen die zu untersuchenden Zellen zunächst eingefroren werden könnten und dann in Richtung der jeweils notwendigen Tests (RNA-basiert, DNA-basiert) aufgearbeitet werden könnten. Dies setzt allerdings auch voraus, dass eine Asservierung des Ausgangsmaterials (gerade bei Knochenmark ist dies von größter Bedeutung, eine erneute Punktion des Patienten ist zu vermeiden) gewährleistet ist. Aufgrund der Vielzahl der möglichen Analyseverfahren ist es deshalb sinnvoll, zunächst zumindest die Befunde der Zytomorphologie und Immunphänotypisierung abzuwarten, bevor größere molekulargenetische Untersuchungen veranlasst werden.
Die aktuelle Diskussion, speziell auch in Bezug auf die Molekulargenetik und das NGS, wird durch die immensen methodischen Fortschritte der letzten Jahre neu zu führen sein. Es stellt sich die Frage, wie molekulargenetische Tests aussehen sollten, um zeitnah, valide und kosteneffektiv durchgeführt zu werden. Dabei erscheint eine Steuerung durch die Standarddiagnostikverfahren von größter Bedeutung [12, 13]. So würde zum Beispiel eine Diagnostik in Richtung akuter Leukämien in der Molekulargenetik sich definitiv unterscheiden von einer Diagnostik in Richtung Lymphome, ebenso sind die myeloproliferativen Neoplasien, inklusive der CMML, in der Molekulargenetik mit einem spezifischen Portfolio adressierbar. Die aktuellen methodischen Möglichkeiten, speziell durch das NGS, machen es aber notwendig zu entscheiden, ob anstelle einer in der Molekulargenetik bisher üblichen Stufendiagnostik einzelner Gene nicht vielmehr eine sogenannte Panel-Diagnostik mit bestimmten Gen-Panels sinnvoller sein könnte. Dies gilt nicht nur im Sinne einer Zeitersparnis, sondern auch bezüglich der entscheidenden molekular getriebenen Beschreibung der jeweiligen Krankheitsentität. Nur dann können eine potenziell zielgerichtete Therapie und die Definition von Markern für die Bestimmung der minimalen Resterkrankung erfolgen. Da die Durchführung von Whole Exome Sequencing oder Whole Genome Sequencing für die Routinediagnostik momentan nicht geeignet ist, könnte die Panel-Diagnostik in der Molekulargenetik in absehbarer Zeit eine zentrale Rolle einnehmen. Die dabei zur Verfügung stehenden Tests sind geeignet, Genmutationen ebenso zu detektieren wie zum Beispiel Fusionsgene zu analysieren. In sehr naher Zeit ist zu erwarten, dass die technischen Möglichkeiten dieses in wenigen Stunden bis Tagen werden abbilden können und dabei Sensitivitäten von 1% oder weniger erreicht werden. Dies wird allerdings für die Routinediagnostik nur anwendbar werden, wenn die erhaltenen Daten mithilfe solider und validierter Software ausgewertet und die Ergebnisse für den behandelnden Hämatologen und seinen Patienten verständlich gemacht werden können.
Zusammenfassung
Aktuell sollte die hämatologische Stufendiagnostik mit dem Phänotyp der zu analysierenden Zellen im peripheren Blut und/oder Knochenmark beginnen. Daneben ist die klinische Anamnese und möglichst eine gezielte Fragestellung des Arztes von grundlegender Bedeutung für die weiteren Analysewege. Ausgehend von diesen Befunden sollten dann eine klassische Chromosomen-Analyse, Fluoreszenz-in-situ-Hybridisierung, Immunphänotypisierung und Immunhistologie oder gegebenenfalls die Molekulargenetik veranlasst werden. Es ist zu beachten, dass für die Primärdiagnostik alle diese Methoden in Kombination sinnvoll sein können, für die notwendigen Verlaufsanalysen (minimale Resterkrankung, MRD) jedoch insbesondere die Immunphänotypisierung und molekulargenetische Methoden geeignet sind.
In der Molekulargenetik hämatologischer Neoplasien hat sich technisch in den letzten Jahren eine Revolution ereignet. Das Wissen über in der Hämatologie veränderte Gene ist dramatisch angestiegen, die methodischen Möglichkeiten zur Analyse haben sich ebenso deutlich erweitert. Es zeichnet sich heute deshalb ein gerader Weg zwischen der Beschreibung des Phänotyps der malignen Zellen über die Zytogenetik hin zur umfassenden Molekulargenetik mit Analyse von sogenannten Gen-Panels ab. Dies ist diagnostisch, therapeutisch und prognostisch bei vielen hämatologischen Systemerkrankungen von grundlegender Bedeutung für Ärzte und Patienten. Der genaue Stellenwert von Hochdurchsatz-Sequenzierungen z. B. im Sinne eines Whole Exome Sequencing muss allerdings noch endgültig definiert werden und bedarf zusätzlich bioinformatischer Expertise. Gerade in Zeiten der sogenannten precision medicine stellt die Definition von molekularen therapeutischen Zielstrukturen und deren Kontrolle als Verlaufsparameter eine zentrale Säule der hämatologischen Diagnostik dar.
(Teil 2 folgt: Möglichkeiten und Grenzen von Next Generation Sequencing (NGS) in der hämatologischen Diagnostik)
Literatur
1. Haferlach T et al. Labordiagnostik bei Leukämien und Lymphomen. 3. Aufl.; Bremen: UNI-MED; 2011.
2. Kohlmann A et al. Perspective on how to approach molecular diagnostics in acute myeloid leukemia and myelodysplastic syndromes in the era of next-generation sequencing. Leuk Lymphoma 2014; 55: 1725-34.
3. Ivey A et al. Assessment of minimal residual disease in standard-risk AML. N Engl J Med 2016; 374: 422-33.
4. Löffler H et al. Hämatologische Erkrankungen. 2. Aufl.; Berlin: Springer; 2012.
5. Shaffer LG et al. ISCN 2013: An international system for human cytogenetic nomenclature. Basel, New York: Karger; 2013.
6. Kern W et al. The role of multiparameter flow cytometry for disease monitoring in AML. Best Pract Res Clin Haematol 2010; 23: 379-90.
7. van de Loosdrecht AA et al. Standardization of flow cytometry in myelodysplastic syndromes: Report from the first European LeukemiaNet working conference on flow cytometry in myelodysplastic syndromes. Haematologica 2009; 94: 1124-34.
8. Cancer Genome Atlas Research Network. Genomic and epigenomic landscapes of adult de novo acute myeloid leukemia. N Engl J Med 2013; 368: 2059-74.
9. Dohner H et al. Acute myeloid leukemia. N Engl J Med 2015; 373: 1136-52.
10. Bejar R et al. TET2 mutations predict response to hypomethylating agents in myelodysplastic syndrome patients. Blood 2014; 124: 2705-12.
11. Matthijs G et al. Guidelines for diagnostic next-generation sequencing. Eur J Hum Genet 2016; 24: 2-5.
12. Godley LA. Profiles in leukemia. N Engl J Med 2012; 366: 1152-3.
13. Malcovati L et al. Diagnosis and treatment of primary myelodysplastic syndromes in adults: Recommendations from the European LeukemiaNet. Blood 2013; 122: 2943-64.

Prof. Dr. med. Dr. phil. Torsten Haferlach
MLL, Münchner Leukämielabor GmbH
Max-Lebsche-Platz 31, 81377 München
+49 89 99017100
+49 89 99017109
www.mll.com
torsten.haferlach[at]mll[dot]com

