Die Zukunft der Massenspektrometrie: Werden Immunoassays bald ersetzt?
DOI: https://doi.org/10.47184/td.2023.03.07Die Massenspektrometrie zeichnet sich u. a. durch eine hohe Selektivität, Sensitivität und Reproduzierbarkeit aus. Es wird nur ein kleines Probenvolumen benötigt und die Verbrauchskosten für die einzelne Messung sind gering. Hinzu kommt ein großes Potenzial für die Multiplex-Diagnostik. Demgegenüber stehen bei den Immunoassays hohe Reagenzkosten und für eine serielle Abarbeitung wird ein relativ großes Probenvolumen benötigt. Die Möglichkeiten zur Multiplex-Diagnostik sind eingeschränkt.
Schlüsselwörter: Flüssigchromatographie, LC-MS/MS, MALDI-TOF, TDM, Neugeborenenscreening
Die Massenspektrometrie (MS) ist ein analytisches Verfahren, bei dem das Masse-zu-Ladungsverhältnis (m/z-Ratio) von Ionen bestimmt wird. Seit Arthur Jeffrey Dempster und Francis William Aston die technischen Grundlagen heutiger Massenspektrometer entwickelt haben – letzterer erhielt dafür 1922 den Nobelpreis für Chemie – ist die Methodik dank zahlreicher Weiterentwicklungen als Standardverfahren in der (bio-)chemischen Analytik nicht mehr wegzudenken.
Der Einsatzbereich der Massenspektrometrie war zunächst (akademischen) Entwicklungslaboratorien vorbehalten. Der Interessenschwerpunkt lag dabei im Bereich „Proteomics“, also der qualitativen und quantitativen Analytik einer Vielzahl von Proteinen und Proteinmodifikationen in Modellsystemen. Die Einsatzmöglichkeiten wurden kontinuierlich auf medizinisch-diagnostische Anwendungsbereiche ausgeweitet, da die Methode aufgrund des physikalischen Analyseprinzips eine hohe Rückführbarkeit zeigt und daher als Referenzmethode gilt.
In der medizinischen Diagnostik hat die Massenspektrometrie heutzutage in folgenden Bereichen eine etablierte Anwendung:
Beim Therapeutischen Drugmonitoring (TDM) wird sie zur präzisen und sicheren Konzentrationsbestimmung von niedermolekularen Arzneimitteln wie Antibiotika oder Immunsuppressiva eingesetzt. Auch hochmolekulare Medikamente wie etwa Biologicals können durch spezielle Probenvorbereitung (tryptischer Verdau) und Identifikation quantotypischer Peptide analysiert werden (Abb. 1).
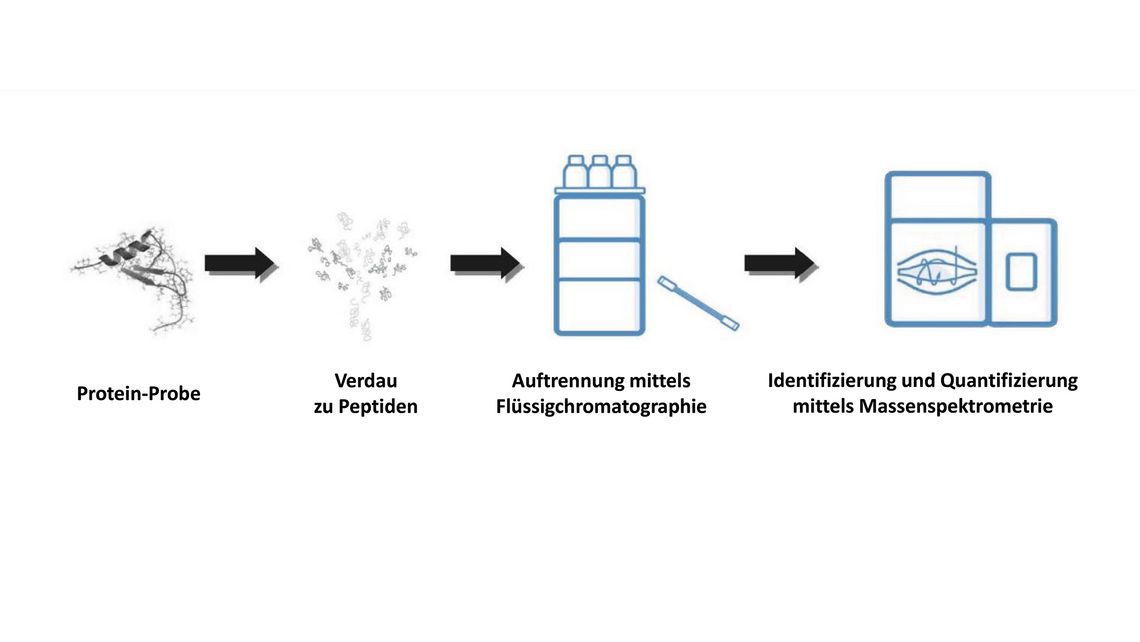
Abb. 1: In einem typischen LC-MS-Arbeitsablauf werden die Proteine aus der Probe extrahiert und durchlaufen entweder einen Anreicherungs-/Abreicherungsschritt vor dem Verdau oder werden einfach durch ein Verdauungsenzym zu Peptiden verdaut. Die Peptide werden anschließend mittels HPLC aufgetrennt, wobei in der Regel eine Umkehrphasensäule verwendet wird. Abschließend erfolgt die Analyse mittels Massenspektrometrie und die Peptide werden mit einer geeigneten Software identifiziert und quantifiziert (Bild: [2]).
Beim Neugeborenenscreening werden entsprechende Metabolite u. a. aus Trockenblutproben quantifiziert, um Stoffwechselerkrankungen zu detektieren.
Die Toxikologie nutzt die hohe Spezifität der Massenspektrometrie in Kombination mit (Flüssig- oder Gas-)Chromatographie (LC bzw. GC) zur eindeutigen Identifikation von Medikamenten und Missbrauchsdrogen im Sinne eines General Unknown Screening. Dabei werden physikochemische Eigenschaften von Inhaltsstoffen einer Probe (Retentionszeit, Muttermasse, Fragmentmasse) mit einer Datenbank abgeglichen, die mit entsprechenden Reinsubstanzen erstellt wurde und heute von mehreren Anbietern kommerziell erhältlich ist.
Die bisher aufgeführten Anwendungen werden in der Regel mit einer LC-MS/MS-Geräteplattform durchgeführt. Das sind Triple-Quadrupol-Massenspektrometer, die mit einer Flüssigchromatographie gekoppelt sind.
Eine weitere, medizinisch-diagnostisch relevante Geräteform ist die MALDI-TOF(matrix-assisted laser desorption ionization time-of-flight)-Massenspektrometrie, die vorzugsweise in der Mikrobiologie zum Einsatz kommt. Hier werden zur Identifikation von Bakterien und Pilzen Proteinmuster erzeugt, die eine hohe Spezies-Spezifität aufweisen. Diese Profile werden mit einer Datenbank aus Referenzkeimen abgeglichen und liefern in Abhängigkeit zum Grad der Übereinstimmung einen Identifikations-Score (ähnlich wie der „Fingerabdruck“ in der Kriminalistik).
Die aktuelle Situation
Der hohe Preis für die außerordentliche Sensitivität und Spezifität der Massenspektrometrie setzt sich aus teurer Hardware, gegebenenfalls aufwendiger Probenaufarbeitung und einem hohen Maß an spezifischem Fachwissen zusammen. Im Falle des Neugeborenenscreenings wird die Problematik der erforderlichen technischen Ausstattung und des hochspezialisierten Fachpersonals dadurch relativiert, dass es in Deutschland durch elf zertifizierte Screeninglabore abgedeckt wird. In der Mikrobiologie hingegen hat die Massenspektrometrie mithilfe benutzerfreundlicher Analyzer und kommerzieller Datenbanken Einzug gehalten.
Derzeit stellt insbesondere beim TDM die hohe analytische Komplexität der Massenspektrometrie eine bedeutende Hürde dar. Gleichzeitig hat die Bestimmung von Medikamentenspiegeln in den letzten Jahren deutlich an Relevanz gewonnen. Dies zeigt sich besonders bei Antibiotika, bei denen mittlerweile verschiedene aktuelle Leitlinien ein Monitoring auch für Arzneimittel mit hoher therapeutischer Breite wie bei β-Laktam-Antibiotika empfehlen. Leider zeigt eine aktuelle Umfrage, dass dies auf deutschen Intensivstationen noch nicht ausreichend umgesetzt werden kann [1]. Derzeit werden TDM-Analysen mittels HPLC-MS/MS (FlüssigchromatographieTandem-Massenspektrometrie) in der Regel als laboratory-developed tests (LDT) durchgeführt, die ein hohes Maß an Fachwissen und spezialisiertem Personal erfordern. Obwohl die gängigen Analyten auch als IVDR-zertifizierte Testkits kommerziell erhältlich sind, wird die Komplexität der Methodik durch die Verwendung dieser Kits nicht spürbar reduziert.
Immunoassays
Heutzutage sind Immunoassays ein Standardverfahren in der klinischen Diagnostik zur Quantifikation spezifischer Antigene oder Antikörper. Sie werden in der Regel als ELISA (Enzyme-linked Immunosorbent Assay) oder CLIA (ChemiLuminescent ImmunoAssay) durchgeführt. Beide Messverfahren erfordern die Bildung eines immunologischen Komplexes aus Analyt und Reagenz für die Messung. Dies führt dazu, dass die Messung sehr empfindlich gegenüber Matrixeffekten ist, insbesondere in Form von unspezifischen Bindungen. Abhängig von der Konzeption des Assays kann das sowohl zu falsch hohen wie auch zu falsch niedrigen Messergebnissen führen. Darüber hinaus ist die Entwicklung der benötigten Antikörper ein kostspieliger und zeitaufwendiger Prozess. Trotz dieser Einschränkungen haben sich Immunoassays aufgrund ihrer einfachen Anwendung bisher in der diagnostischen Routine behauptet. Multiplexing ist aufgrund von schwer absehbaren Antikörperinteraktionen nur mit sehr viel Aufwand umsetzbar, sodass für jede Messung ein eigener Ansatz mit je einem Probenvolumen in der Größenordnung von 10–200 µl benötigt wird. In der klinischen Diagnostik fällt dabei jedoch hauptsächlich der erhöhte Aufwand durch die mehrfachen Ansätze als Schwäche ins Gewicht.
Massenspektrometrie
Im Gegensatz zu Immunoassays basiert die Detektion von niedermolekularen Substanzen oder Proteinen mittels Massenspektrometrie auf einem grundlegend anderen Prinzip. In der Regel wird das Verfahren der HPLC-MS/MS angewendet. Dabei wird die zu messende Probe zunächst chromatographisch aufgetrennt. Nach einer methodenspezifischen Zeit gelangen die jeweiligen Analyten durch Elektrospray-Ionisation in den ersten Massenfilter, in dem sie basierend auf ihrer spezifischen Masse selektiert werden. Da verschiedene Moleküle durchaus identische Massen haben, werden die Analyten in einer Kollisionszelle mittels Stoßionisation fragmentiert. Dadurch entstehen substanzspezifische Fragmente, die in einem zweiten Massenfilter analysiert und quantifiziert werden. Durch die zeitliche Trennung der Analyten mittels Chromatographie können aus einem sehr geringen Probenvolumen von etwa 10 µl eine Vielzahl von Analyten aus einem Ansatz bestimmt werden. Gleichzeitig zeichnet sich die Methode durch hohe Sensitivität, hohe Spezifität und hohe Reproduzierbarkeit aus. Die größte Schwäche besteht auch hier wieder in den Matrixeffekten. Insbesondere die Gegenwart hochabundanter Proteine wie Albumin im Blutplasma kann die Messung niedrig konzentrierter Substanzen stören. Bei der Messung von niedermolekularen Substanzen wie beim TDM kann eine Proteinpräzipitation oder -filtration Abhilfe schaffen. Die Bestimmung von Proteinen erfordert jedoch eine komplexe Probenvorbereitung zur Abreicherung abundanter „Hintergrund“-Proteine oder Anreicherung von Zielproteinen. Zudem können größere Proteine erst nach proteolytischer Spaltung massenspektrometrisch gemessen werden. Es gibt jedoch Fortschritte in diesem Bereich, die eine zunehmende Automatisierung ermöglichen. Insbesondere die Verwendung mehrerer LC-Säulen (sogenannter Trap-Säulen) oder die Verwendung magnetischer Partikel sind hier zu erwähnen [2, 3]. Auch posttranslationale Modifikationen von Proteinen und damit ggf. bestimmte Aktivitätszustände sind mit Massenspektrometrie detektierbar. Die präzise Quantifizierung von aktiven Biomolekülen ist auch für das TDM und andere Fragestellungen wie dem Vitamin D- oder Steroidhormonhaushalt von großer Bedeutung. Dieser Mehrwert kann derzeit nicht durch Immunoassays abgebildet werden (Tab. 1).
Tab. 1: Stärken und Schwächen von Massenspektrometrie und Immunoassays.
Massenspektrometrie | Immunoassays |
|---|---|
Stärken | Stärken |
Hohe Selektivität Hohe Sensitivität Hohe Reproduzierbarkeit Geringe Kosten pro Messung Multiplexing Niedriges Probenvolumen | Einfache Anwendung Günstiges Equipment |
Schwächen | Schwächen |
Teures Equipment Komplexe Messung und Auswertung | Antikörperbasiert Hohe Reagenzkosten Eingeschränktes Multiplexing Hohes Probenvolumen |
Was bringt die Zukunft?
Es ist unwahrscheinlich, dass Immunoassays in absehbarer Zeit vollständig durch Massenspektrometrie ersetzt werden. Beide Techniken haben ihre spezifischen Vor- und Nachteile. Immunoassays werden vor allem in kleineren (Krankenhaus-)Laboratorien weiterhin ein wichtiger Bestandteil der klinischen Routinediagnostik bleiben, wenn man beispielsweise an die Bestimmung von TSH oder Troponin im Setting einer Notaufnahme denkt.
Die Automatisierung gilt als ein wichtiger Faktor bei der Verbreitung der Massenspektrometrie in medizinisch-diagnostischen Laboratorien. Beim TDM etwa würde ein höherer Automatisierungsgrad und damit eine leichtere Verfügbarkeit der Methodik zu einer Ergänzung und teilweisen Substitution von Immunoassays durch die Massenspektrometrie führen.
Ein vollautomatisierter LC-MS/MS-Workflow ist aktuell nicht kommerziell verfügbar. Auf der Euromedlab 2023 in Rom wurde die Einführung einer vollautomatisierten klinischen Massenspektrometrie für das kommende Jahr angekündigt. Neben Medikamenten sollen dann auch andere „small molecules“ wie Missbrauchsdrogen, Stoffwechselprodukte, Vitamine oder Peptidhormone analysiert werden können.
Auch höhermolekulare Zielstrukturen wie etwa freie Leichtketten mit einem Molekulargewicht von ~25 kDa können mit MALDI-TOF-MS für die Myelomdiagnostik nutzbar gemacht werden. Der Nachweis von klonotypischen Leichtketten hat gegenüber den Standardverfahren wie Immunfixationselektrophorese eine deutlich höhere Sensitivität und Spezifität. In laufenden Studien wird geprüft, welchen diagnostischen Stellenwert dieses neue Verfahren etwa bei dem longitudinalen Monitoring oder der Detektion einer minimalen Resterkrankung haben wird.
Schließlich wird in absehbarer Zeit auch die Analytik von Proteinen durch MS-basierte Techniken immer besser abbildbar sein. So ist etwa die zielgerichtete Konzentrationsbestimmung einzelner Proteine wie etwa therapeutischer Antikörper in einzelnen Laboratorien schon als LDT umgesetzt. Grundvoraussetzung hierfür ist die Verfügbarkeit von Stabil-Isotopen markierten (SIL) internen Standards, die kommerziell verfügbar sind.
Eine hochkomplexe, multiparametrische Quantifizierung von hunderten Proteinen aus klinischem Material ist heute bereits ebenfalls technisch und gut reproduzierbar möglich. Auch hier gibt es schon erste kommerzielle Anbieter. Allerdings sind solche Anwendungen noch auf translationale Forschungsprojekten oder klinische Studien beschränkt.
Fazit
Zusammenfassend bleibt festzuhalten, dass sich die beiden Techniken heute schon sinnvoll ergänzen und voraussichtlich kontinuierlich technisch weiterentwickelt werden, um den Anforderungen verschiedener Anwendungsgebiete zukünftig besser gerecht werden zu können. Die Entscheidung für den Einsatz einer bestimmten Methode hängt von spezifischen Anforderungen, ökonomischen und logistischen Faktoren ab.

