Schnellere Erregeridentifizierung bei Meningitis
Liquordiagnostik mithilfe von MALDI-TOF MS-ID
Die MALDI-TOF-Massenspektrometrie ist ein innovatives Verfahren, das bei Verdacht auf Meningitis mit hoher Verlässlichkeit Bakterien aus CSF-Kulturen identifizieren kann und somit dem behandelnden Arzt therapierelevante Informationen ca. 36 Stunden früher als die herkömmliche Labordiagnostik zur Verfügung stellt.
Schlüsselwörter: bakterielle Meningitis, Massenspektrometrie, MALDI-TOF MS-ID, Blutkultur
Die bakterielle Meningitis gehört zu den lebensbedrohlichen Notfällen im Krankenhaus. Ihre Inzidenz liegt in westlichen Industrieländern bei etwa drei bis sechs Fällen pro 100.000 Einwohner [1] und die Letalität ist abhängig vom auslösenden Erreger sehr hoch. Für Meningokokken liegt sie bei 3 bis 10%, für Pneumokokken und Listerien sogar bei 20 bis 40% [2, 3]. Obwohl Kinderimpfungen gegen Meningokokken und Pneumokokken sowohl zu einem Anstieg des Erkrankungsalters als auch zu einem Abfall der Inzidenz geführt haben, ist die schnelle Diagnostik weiterhin der Grundstein einer möglichst frühzeitigen, erfolgreichen Meningitis-Therapie [4, 5].
Die klassischen Symptome der bakteriellen Meningitis wie Fieber, Nackensteife und verändertes Bewusstsein finden sich nur in der Hälfte der Fälle und sind somit kein zuverlässiges Kriterium [6]. Deshalb nimmt die Laboratoriumsdiagnostik und hier vor allem die Liquoranalytik nach Lumbalpunktion bei der Diagnosestellung eine Schlüsselrolle ein. Zur schnellen Abklärung hat sich eine Kombination aus klinisch-chemischer Analyse (Totalprotein, Glukose und Laktat) und Zelldifferenzierung bewährt. Die mikrobiologische Erregerisolation mit anschließender Identifikation und Resistenztestung ist obligat für eine zielgerichtete Antibiotika-Therapie. Allerdings können Antibiotikaresistenzen zu einem Versagen der initialen Breitbandantibiose führen und sich negativ auf den Outcome des Patienten auswirken [7].
Praktisches Vorgehen
Zum Erregernachweis und für seine Isolation beimpft und inkubiert man für jeden Patienten mit klinischem Meningitisverdacht eine aerobe Blutkulturflasche mit Cerebrospinalflüssigkeit (CSF), sowie zwei Pärchen Blutkulturen mit peripherem Blut (vgl. Abb. 1). Die CSF-Kultur wurde vorher zur Wachstumsverbesserung anspruchsvoller Keime, wie zum Beispiel Hämophilus influenzae, mit NAD und Hämin supplementiert (FOS, Becton Dickinson). An die Probengewinnung schließt sich eine empirische Antibiotika-Therapie mit begleitender hochdosierter Glukokortikoidgabe unverzüglich an.
Parallel analysiert man die CSF-Proben durchflusszytometrisch sowie klinisch- chemisch und fertigt ein Cytospin-Präparat an. Da die Sensitivität eines Liquorpräparates zum Bakteriennachweis nicht sehr hoch ist, kann auf die Kultur zur Erregeridentifikation nicht verzichtet werden [8].
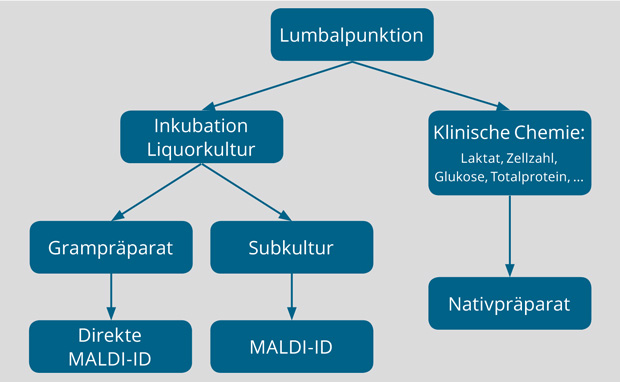
Für eine klassische mikrobiologische Erregeridentifikation muss die Kulturflüssigkeit, nachdem sie als positiv gemeldet wurde, auf verschiedenen Festplattenmedien subkultiviert werden.
Experimentelle Studie
Um eine möglichst schnelle Erregeridentifikation anbieten zu können, haben wir einen Assay auf Basis der MALDI-TOF-MS entwickelt, der rund um die Uhr durchführbar ist und CSF-Kulturen ohne vorhergehende Subkultivierung verarbeiten kann. Die Extraktion und Identifikation der positiven Kulturflaschen erfolgte aufgrund der personellen Situation zweimal täglich um 6:00 und 20:00 Uhr.
In dieser Studie beimpften wir bei klinischem Verdacht auf eine bakterielle Meningitis eine aerobe Blutkulturflasche (BacTec Plus™, BD®) nach der Lumbalpunktion mit ca. 5 ml nativer CSF. Die Flaschen wurden automatisch inkubiert, bis sie durch das System als positiv gemeldet wurden.
Vor der massenspektrometrischen Analyse untersuchten wir von jeder positiven CSF-Kultur zuerst ein Grampräparat mikroskopisch, um polymikrobielle Infektionen auszuschließen. Anschließend wurde aus der CSF-Kulturflasche unter einer Laminar-Flow-Werkbank und sterilen Bedingungen 1 ml Kulturflüssigkeit entnommen. Die Zellen der Kultur wurden mit TritonX-100 (Sigma-Aldrich®) lysiert, pelletiert und mit 0,9-prozentiger NaCl-Lösung gewaschen. Nach dem Abzentrifugieren und Verwerfen des Überstandes konnten wir die bakteriellen Proteine mithilfe von Ameisensäure und Acetonitril extrahieren. Die Lösung wurde auf ein MALDI-Target aus Stahl aufgetragen und mithilfe einer HCCA-Matrix ionisiert. Die Analyse erfolgte automatisiert durch das Programm Biotyper (Vers. 3.0, Bruker Daltonics®), wobei jede Probe insgesamt viermal aufgetragen wurde. Bei jeder Analyse wurden zwei E.-coli-Kontrollen (ATCC® 25922™) mitgemessen.
Der Abgleich des Summenspektrums mit der Referenzdatenbank ermöglicht sowohl eine Identifikation des Keims als auch eine Bewertung des Ergebnisses (Score). Eine valide Identifikation auf Genus-Ebene nehmen wir ab einem Score von 1,8 und darüber an, wenn dieser auf mindestens zwei Spots erreicht wurde. Entsprechend beurteilen wir ein Ergebnis von 2,0 und darüber als Identifikation auf Speziesebene.
Ergebnisse
Die Analyse klinischer Primärproben (n = 67) ergab in 82,7% der Fälle eine sichere Identifikation; der erzielte Score lag bei 2,00 ± 0,205. Ein Vergleich mit den Identifikationsergebnissen von subkultivierten Keimen zeigte, dass es in keinem einzigen Fall zu einer Falschidentifikation kam.
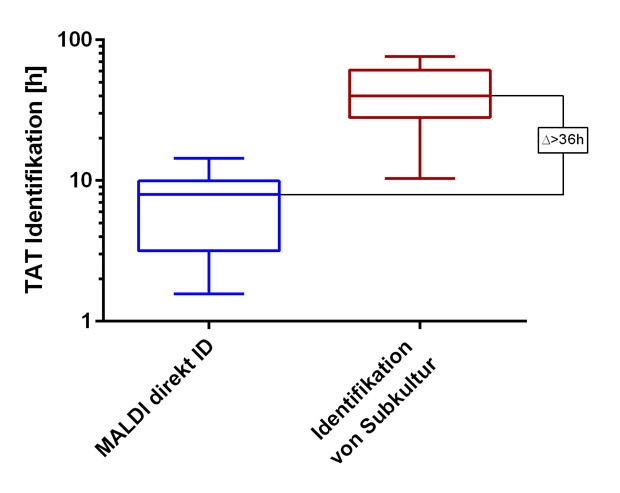
Im Vergleich zur Identifikation aus Subkulturen mit Vitek2® oder MALDI-TOF-MS erzielten wir eine Zeitersparnis von ca. 36 h bis zur Identifikation: Während die Direktextraktionsergebnisse nach 7,26 ± 4,06 h vorlagen, dauerte die Analyse in der Subkultur 43,30 ± 1,86 h (Abb. 2).
Diskussion
Wir konnten somit zeigen, dass es technisch möglich ist, mithilfe der MALDI-TOF-MS eine bakterielle Identifikation von CSF-Kulturen rund um die Uhr durchzuführen. Dies eröffnet dem behandelnden Arzt die attraktive Möglichkeit, eine empirische Antibiotika-Therapie schneller als bisher keimgerecht anzupassen. So zeigt eine aktuelle Cochrane-Analyse, dass nur Patienten mit einer Pneumokokken-Meningitis von der zusätzlichen Therapie mit Kortikosteroiden (Dexamethason) profitieren. Für eine Infektion mit Staphylococcus aureus wird dagegen empfohlen, die antimikrobielle Therapie um Fosfomycin, Rifampicin oder Linezolid zu erweitern [9].
Eine Limitation des beschriebenen Ansatzes ist die mögliche abnahmebedingte Kontamination der Liquorkultur mit Hautflora. Dies führt dazu, dass kein valides Summenspektrum erzeugt werden kann und keine Identifikation gelingt. Da der Assay eine bakterielle Dichte von mehr als 103 CFU/ml voraussetzt, erfordert das Verfahren eine zeitaufwendige, kulturelle Anreicherung der Bakterien.
Testsysteme, die auf molekulargenetischen Methoden, insbesondere auf der PCR basieren, leiden an dieser Einschränkung nicht. Sie weisen bakterielle oder virale Nukleinsäuren innerhalb weniger Stunden im Liquor nach und liefern somit schnellere Resultate als das hier vorgestellte massenspektrometriebasierte Verfahren. Allerdings spiegeln sie nicht unbedingt die reale Infektionssituation wider. So führt zum Beispiel eine Amplifikation nicht-vitaler bakterieller Nukleinsäuren zu falsch-positiven Befunden.
Dazu kommen bei molekularbiologischen Verfahren die noch immer hohen Kosten der Systeme, die eine breite Anwendbarkeit limitieren. Nicht immer gelingt der Sensitivitätsnachweis mit molekularbiologischen Verfahren, sodass dann für die Resistenztestung weiterhin eine Kultur benötigt wird.


